生工技术 | SNP的检测技术——LDR 连接酶法
在介绍这种方法之前,需要先了解一下Taq DNA连接酶,当蓝色和紫色两条单链与同一条DNA模板互补配对时,只有他们之间完全没有缝隙的时候,TaqDNA连接酶才能将其连接成为完整的一条链。
当蓝色和紫色链的拼接处有单个碱基不匹配时,会导致其末端翘起,这种情况下Taq DNA连接酶是无法进行连接作用的,这样变性之后,仍会保持两条单链的形式。
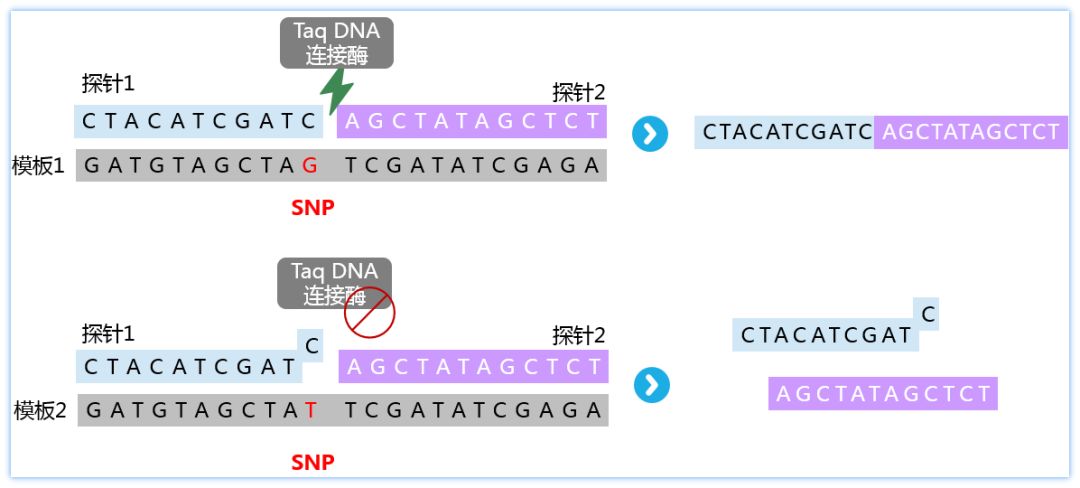
从上方示意图汇总可以看出,DNA模板仅有一个碱基不同,就会导致连接产物的完全不同。因此,这种方法可以用来检测SNP的不同基因型。
具体如何检测出来的呢?假如这里有三个SNP位点,A、B和C,针对每个SNP位点的基因型,分别设计两套探针,每一套探针由蓝色的可变长度链和粉色的荧光标记链组成。例如针对SNP A,我们设计probe set1和2,分别检测基因型A和T。如果SNP位点基因型是A,那么probe set 1可以完全与模板互补配对,从而被连接到一起。如果也有T基因型,那么probe set 2也可以被连接到一起。
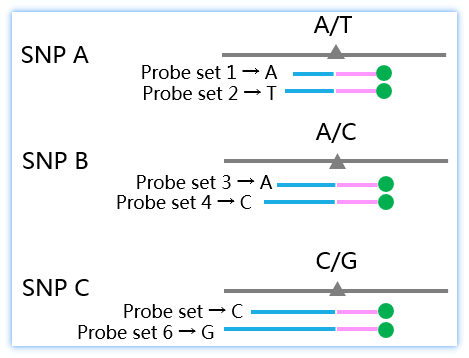
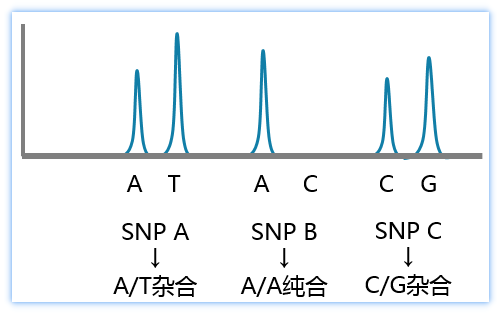
大家同时可以注意到,所有这些probe set的长度均是不一样的,从而在毛细管电泳中能够进行区分不同的长度,这样,可以将三个SNP的链接体系放到一起进行检测,从而大大降低了成本。从右侧结果示意图中可以轻松判断出来,SNP A位点的基因型是A/T杂合,B位点是AA纯合,C位点则是CG杂合。
PCR-LDR连接酶法的技术流程包括以下几个部分1. 多重PCR扩增:获得目标位点所在的片断;2. 多重LDR连接:得到长度各异的检测产物;3. 将连接产物进行毛细管电泳检测:得到不同位置的不同峰值;4.根据SNP峰形结果,进行数据分析,判断SNP的基因型。
这种方法有很多优点,:
一是通用性强:适用各种SNP位点的分型,
二是分型准确,检出率高,
三是检测快速,跟其他方法相比,检测条件更易控制,从DNA样本到数据的产出只要几周。
但是这种方法对于位点的通量比较低,因为所有的连接产物需要区分开长度,并在同一根毛细管中进行检测,限于毛细管电泳适合的DNA长度,SNP位点不能太多(通常为10个以内),否则就只能分成两批去进行毛细管电泳了。
生工生物
LDR 连接酶法SNP检测服务
Taqman探针的检测原理是利用Taq DNA聚合酶的5-3核酸外切酶活力对探针本身进行酶切 。Taqman探针的两端分别标记有荧光基团和对应的淬灭基团。当探针完整时,oligo相当于一条锁链将荧光基团和淬灭基团紧紧拴在一起,导致荧光基团发出的光被淬灭基团吸收。当探针被Taq酶切断之后,荧光基团得以释放从而导致荧光值上升。荧光值的变化会被荧光定量PCR仪或者其他检测仪器记录下来。
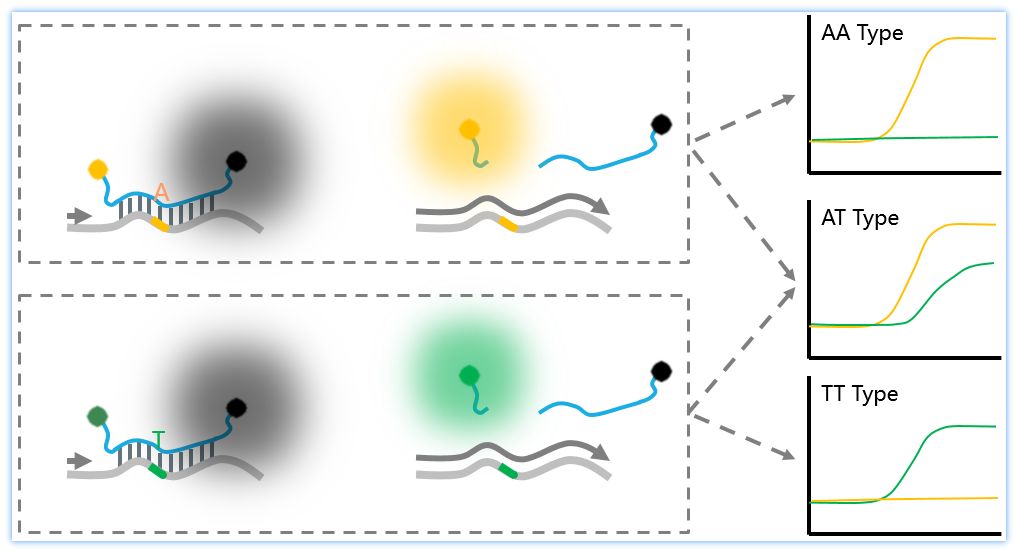
MGB探针非常短,仅有十几个核苷酸的长度,针对不同的SNP基因型,分别设计其对应的探针,不同的探针带有不同的荧光基团,在PCR过程中,只有与模板完全匹配的探针才能完全互补配对,并被水解发光。哪怕只有SNP位点的一个碱基不同,也会导致探针无法结合。因此,根据PCR之后的扩增曲线,即可判断出样本的基因型。
Taqman-MGB探针法 的操作流程仅需要以提取的DNA为模板,加入引物及位点特异性探针,去进行荧光定量PCR扩增,从扩增曲线或SNP分型象限图中就可以判断出样本的基因型。
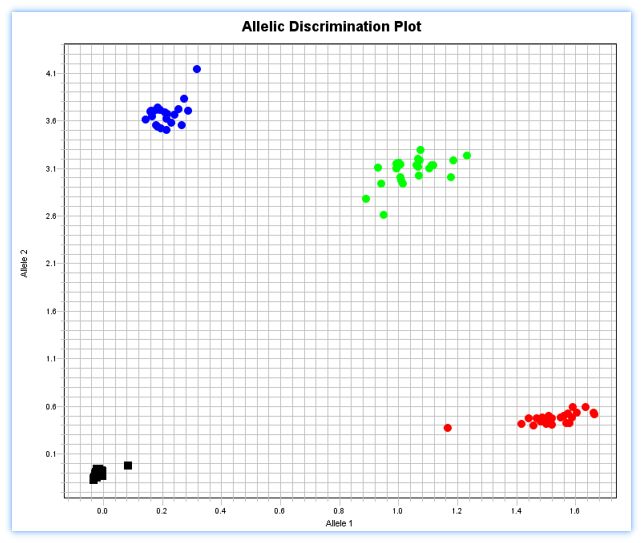
从流程图以及结果示例上看,Taqman-MGB探针法操作简便,得到基因组DNA之后直接就可以检测,无需做其他处理。结果也非常清晰容易判读。目前多数科研和生产机构都配备了荧光定量PCR仪器,技术门槛比较低。尤其适用于针对某个关键多态性位点开发相应的诊断试剂盒。
但是,这种方法不适合一次性检测多个位点,针对每一个SNP位点都要涉及对应的引物和探针,探针合成费用比较高。
生工生物
MGB探针法SNP检测服务
service3@sangon.com

