测序知多少之MeRIP-seq
随着高通量技术的发展,转录组测序也愈发成熟,与此同时,更多的研究不满足于只关注遗传学方向,表观遗传乃至于RNA的表观遗传,越来越多的进入到研究者的视野中。在RNA甲基化测序中,研究较多的就是m6A转录组测序,MeRIP-seq。
什么是m6A
要知道什么是MeRIP-seq,我们就要先了解它所检测的m6A是什么。m6A是指腺嘌呤(A)的第6位氮(N)原子上发生的甲基化修饰。N6-甲基腺苷(m6A)是高等真核生物中mRNA和长链非编码RNA(lnRNA)上最普遍的修饰[1]。主要发生在RRACH序列(其中R=A或G,H=A,C或U)中[2,3],在终止密码子附近、3'非翻译区(3'UTR)和长内部外显子中富集[4,5]。在细菌和病毒的RNA中也发现了m6A的存在[6,7]。m6A可以调节RNA成熟、剪切、转运、降解及翻译等代谢过程,在分子、细胞和生理水平上有助于实现多种基本生物学功能。
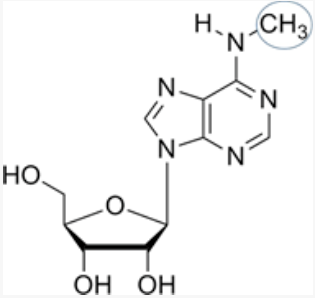
图1 N6-甲基腺苷(m6A)修饰
m6A的功能
自从1948年发现了DNA核苷酸中的第一种修饰[8],表观遗传学的概念被大众所熟知,随着后来研究的深入,表观遗传学通过调节基因表达在细胞功能中的作用变得明显起来。与DNA和蛋白质不同,RNA曾被认为不太重要,被认为仅仅是连接DNA中储存的信息和合成的蛋白质之间的一个过渡元素[9]。直到20世纪80年代,人们才开始了解RNA分子,不仅仅是编码肽的功能。到目前为止,已经在不同类型的RNA中发现了100多种修饰[10]。这些修饰调节着RNA代谢和相关生理过程的几乎所有方面,使它们成为转录后基因调节的关键组成部分[9,11]。
m6A的调节因子可以分为3类:甲基化转移酶(writers)、去甲基化酶(erasers)和m6A阅读蛋白(readers)。甲基化转移酶复合物(MTC)催化m6A的甲基化过程,也被称为“写入者(writers)”。去甲基化酶,也称为“擦除者(erasers)”,可以去除m6A。m6A阅读蛋白可以识别m6A,与mRNA结合并发挥相应的功能(图2)。甲基化转移酶、去甲基化酶和m6A阅读蛋白之间的相互作用与癌症的发生发展密切相关[12]。
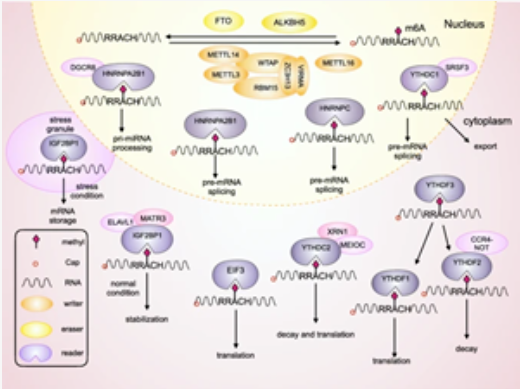
图2 m6A的机制[12]
已有研究证明m6A甲基化修饰在转录后调控中发挥作用,在调控基因表达、剪接、RNA编辑、RNA稳定性、控制mRNA降解、介导环状RNA翻译等方面扮演重要角色,这也意味着mRNA甲基化参与了多种生物学过程,如细胞分化、生物节律等,在包括肿瘤、肥胖和不育等疾病的发生发展中的作用也有报道,因此对RNA m6A甲基化修饰的研究具有重要意义。m6A修饰失调与多种人类疾病有关,因此它在各种癌症和疾病的治疗中具有潜在的治疗应用。免疫系统对于抵抗感染和癌症至关重要。
MeRIP-seq
MeRIP-seq(甲基化RNA 免疫沉淀结合高通量测序)是研究细胞内转录组表观修饰的关键技术,是一种在转录组层面研究细胞内 mRNA 以及 lncRNA甲基化定位的技术,可高精度地检测全转录组范围内的RNA 甲基化。其原理是基于m6A特异性抗体识别并结合RNA上m6A,以免疫共沉淀方法富集片段,并配合高通量测序技术,从而在转录组范围内研究发生甲基化的RNA区域。
meRIP-Seq是目前分析m6A修饰分布的最常用检测方法,可以高效的获得实验结果,对于深入研究RNA甲基化修饰在疾病发展中的功能有重要作用。
生工推荐
生工生物工程(上海)股份有限公司自2011年成立高通量测序部以来,逐步引进了illumina,Life,DNBSEQ,PacBio、Nanopore、等二代/三代测序平台。建立了2000平米高标准实验室(通过了国家ISO认证)并搭建了50 PB级存储及运算能力的服务器集群,能满足绝大多数科研客户的研究需要。同时,生工高通量测序团队建设也在不断完善中,目前有100余专职员工,其中高级技术人员以及高级生物信息工程师占比50%以上,10年来,通过生工高通量测序服务所发表的文章累计影响因子在10000分以上。
除了RNA甲基化项目,我们9月甲基化/羟甲基化多平台促销,也强势来袭。
如果各位老师对我们表观遗传学测序项目感兴趣的话,欢迎致电与我们技术支持沟通。

参考文献
1.Raghda A. Elsabbagh, Mona Rady, Carsten Watzl, Khaled Abou-Aisha & Mohamed Z. Gad. Cell Communication and Signaling volume 20, Article number: 140 (2022).
2.Bokar JA, Shambaugh ME, Polayes D, Matera AG, Rottman FM. Purification and cDNA cloning of the AdoMet-binding subunit of the human mRNA (N6-adenosine)-methyltransferase. Rna. 1997;3:1233–47.
3.Wei CM, Moss B. Nucleotide sequences at the N6-methyladenosine sites of HeLa cell messenger ribonucleic acid. Biochemistry. 1977;16:1672–6.
4.Dominissini D, Moshitch-Moshkovitz S, Schwartz S, Salmon-Divon M, Ungar L, Osenberg S, Cesarkas K, Jacob-Hirsch J, Amariglio N, Kupiec M, et al. Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq. Nature. 2012;485:201–6.
5.Meyer KD, Saletore Y, Zumbo P, Elemento O, Mason CE, Jaffrey SR. Comprehensive analysis of mRNA methylation reveals enrichment in 3′ UTRs and near stop codons. Cell. 2012;149:1635–46.
6.Deng X, Chen K, Luo GZ, Weng X, Ji Q, Zhou T, He C. Widespread occurrence of N6-methyladenosine in bacterial mRNA. Nucleic Acids Res. 2015;43:6557–67.
7.Desrosiers R, Friderici K, Rottman F. Identification of methylated nucleosides in messenger RNA from Novikoff hepatoma cells. Proc Natl Acad Sci U S A. 1974;71:3971–5.
8.Hotchkiss RD. The quantitative separation of purines, pyrimidines, and nucleosides by paper chromatography. J Biol Chem [Internet]. 1948;175(1):315–32.
9.Jacob R, Zander S, Gutschner T. The dark side of the epitranscriptome: Chemical modifications in long non-coding rnas. Int J Mol Sci. 2017;18(11):2387.
10. Liu C, Yang Z, Li R, Wu Y, Chi M, Gao S, et al. Potential roles of N6-methyladenosine (m6A) in immune cells. J Transl Med [Internet]. 2021;19(1):251.
11.Zhao BS, Roundtree IA, He C. Post-transcriptional gene regulation by mRNA modifications. Nat Rev Mol Cell Biol. 2016;18(1):31–42.
12.Liuer He, Huiyu Li, Anqi Wu, Yulong Peng, Guang Shu & Gang Yin. Functions of N6-methyladenosine and its role in cancer. Molecular Cancer. 2019; 18: 176.
.png)

