生工技术 | 使用NUPACK分析核酸的二级结构
使用NUPACK在线网站分析核酸二级结构
在拿到核酸序列时,我们经常会有序列二级结构的分析需要。比如说一条DNA是否会形成发夹结构,两条DNA之间在什么温度下会形成稳定的双链等类似的需求。小编之前使用过Mfold,RNAstructureWeb等在线网站用于分析单条DNA序列的二级结构,但是对于多条DNA形成的二级结构,上述提到的在线网站就有些力不从心了。在这里小编介绍一个在文献中使用频率较高的在线网站NUPACK,这个软件没有Mfold,RNAstructureWeb那么亲民,很多设置的地方需要自己手动尝试并结合注释去理解。
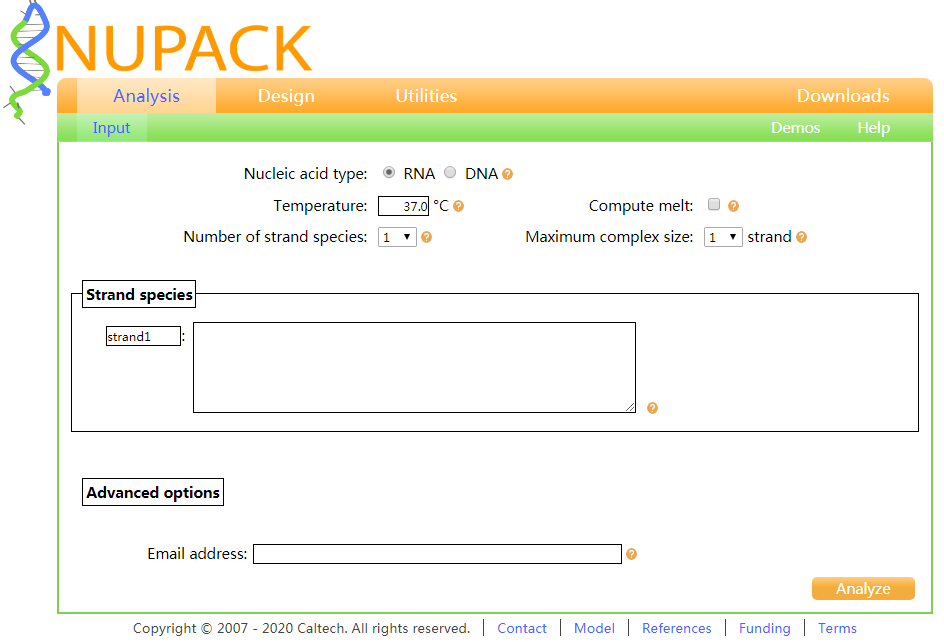
NUPACK软件分析的基本设置
NUPACK在线网站的网址是(http://www.nupack.org/),进入网站后,我们首先点击Analysis进行序列分析页面。
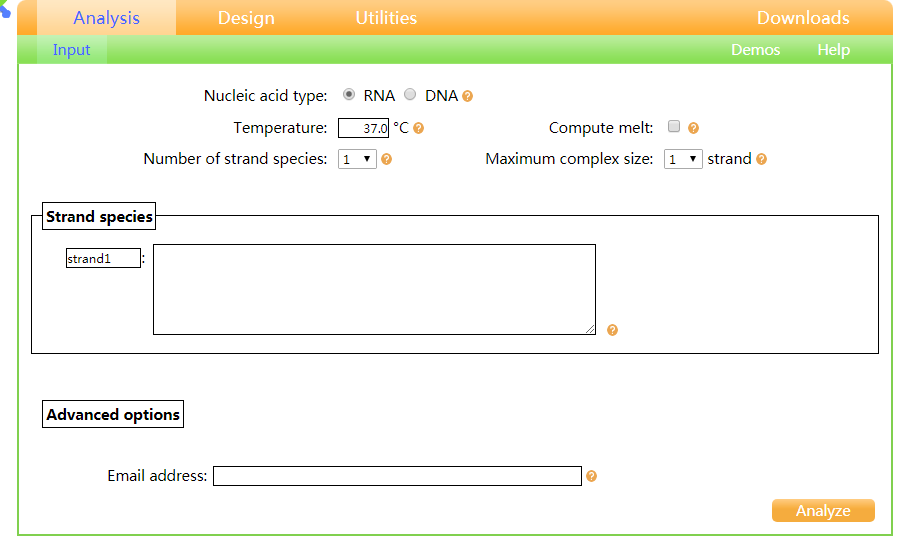
在这个界面我们需要做的是输入DNA的序列,并进行各种参数的设置。
首先是Nucleic acid type,根据分析序列的核酸类型选择RNA或者DNA。
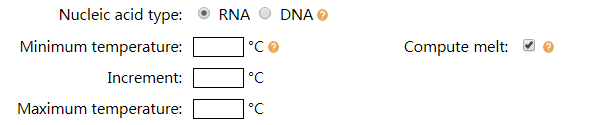
接下来是Temperature,代表了需要分析核酸序列结构所处的环境温度,这里默认是37℃。Compute melt,在这个选项上打钩之后,可以分析一定温度范围内的核酸序列二级结构。勾选之后可以进行三个参数的设置,Minimum temperature,分析的起始温度,一般设置为靠近室温的20 ℃或者25 ℃;Maximum temperature,分析的最高温度,根据自己的需求设定,可以设置在70-95 ℃。Increment,温度的刻度,一般设置为整数的5 ℃或10 ℃。比如说,我们把起始温度设置20 ℃,最高温度设置在40℃,刻度设置为5 ℃,这样我们就可以在20 ℃,25 ℃,30 ℃,35 ℃和40 ℃这5个温度下分析核酸序列的二级结构。
Number of strand species这个下拉框可以选择需要分析的核酸序列的个数。比如说分析一条DNA链是否能形成发夹,就选择1。两条DNA链是否能够形成二聚体,就选择2,以此类推。
Maximum complex size这个代表分析的二级结构所包含的核酸序列的数量,默认值为1。一条DNA,它可能会形成发夹结构这一种二级结构,如果我只需要知道发夹的情况,Maximum complex size这里设置为1就行了。但是如果我们还想了解这条DNA是否会两两形成自身的二聚体结构,或者想要知道还有多少的单链DNA没有形成发夹,Maximum complex size就可以设置为2。
![]()
要注意的是,一旦Maximum complex size大于等于2,那么就要在Strand species里对DNA链的浓度进行设定。在分析结果中也会显示多种结构各自的浓度信息。Maximum complex size这里的数值绝不是越大越好,数值越大,分析的时间也会变得比较漫长,一般设置为2-3即可。Strand species,这里的主要功能是输入核酸序列,也可以输入核酸序列的浓度,多条核酸序列的浓度都可以单独进行设定。
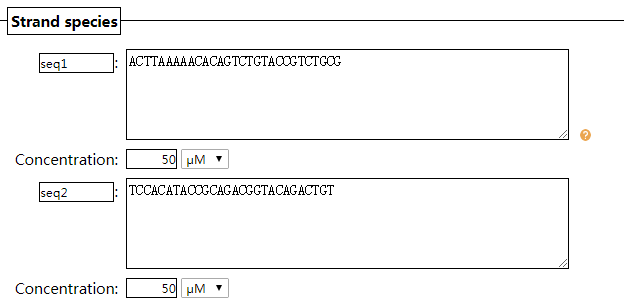
点击Advanced options可以进行更多参数的设定,RNA有两篇参考文献的计算方法可以选择,而DNA只有一篇文献,就是经典的SantaLucia, 1998。小编的日常工作分析主要是DNA,所以这里按照默认的那篇文献即可。Dangle treatment,这里是计算未配对的DNA碱基对二级结构能量的影响,选择默认的some即可。然后我们可以对钠离子Na+和镁离子Mg2+进行设定,Na+的设定范围在0.05~1.1 M,Mg2+的设定范围在0~0.2 M。Specify additional ordered complexes在这里输入想要定制的那种特殊的核酸序列之间形成的二级结构,比如在想要知道输入的三条核酸序列中的第一条和第三条和第二条形成的二级结构,那么就输入1 2 3 2。通常Specify additional ordered complexes这个位置空着不填即可。Email address在这里可以设置接收分析结果的邮箱,当然空着在线观看分析结果也是可以的。

看完这一系列的设定,是不感觉界面冷冰冰,还有点发懵?小编刚开始也是这样的感觉,没关系,下面进入实例分析环节。相信看过实例分析,大家会开始喜欢这个软件,因为它的功能性实在是完爆之前提到的Mfold和RNAstructureWeb。
案例1:
单条DNA的发夹结构分析
以CCTCCCTAAGACTCAGGGAA这条DNA序列为例,我们来分析单条DNA的发夹结构。小编所用的参数设置参考下图,
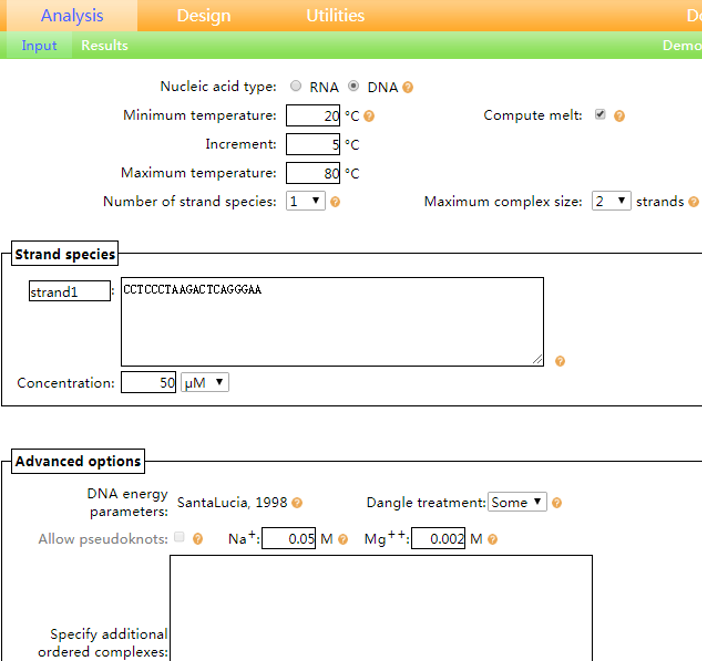
设置完毕后,点击网页右下角的Analyze,进行结果页面。
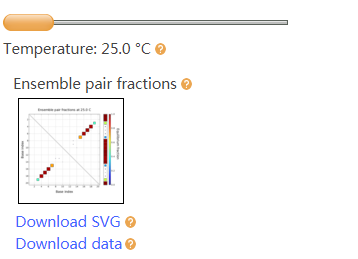
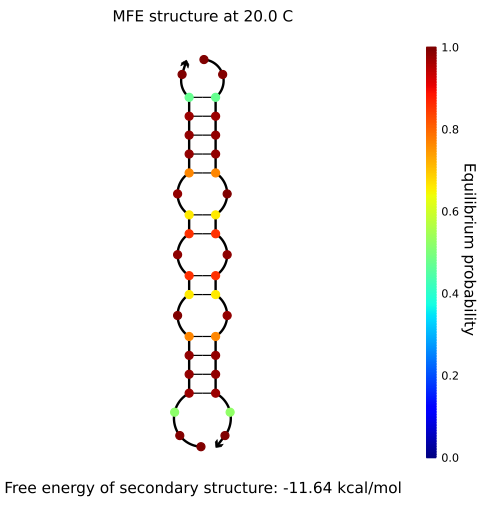
在结果页面,小编的第一感觉是:我想要的高级结构的图在哪?别慌,在Equilibrium concentrations平衡浓度一栏,已经出现了两个DNA结构,一种是DNA自身形成的二级结构,另一种是两条DNA之间形成的二级结构。
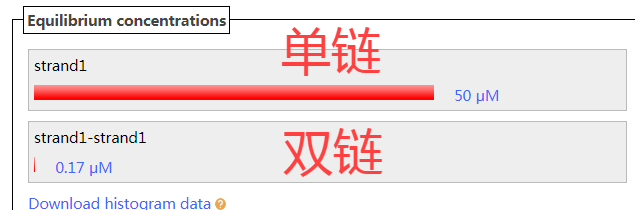
这里还显示了两种结构的浓度,分别为50 uM和0.17 uM。之所以两种二级结构的浓度相加大于我们设置的DNA链的浓度,是因为二级结构的浓度进行了四舍五入的关系。我们点击Equilibrium concentrations的第一个灰色区域即可进入第一个二级结构的信息页面。
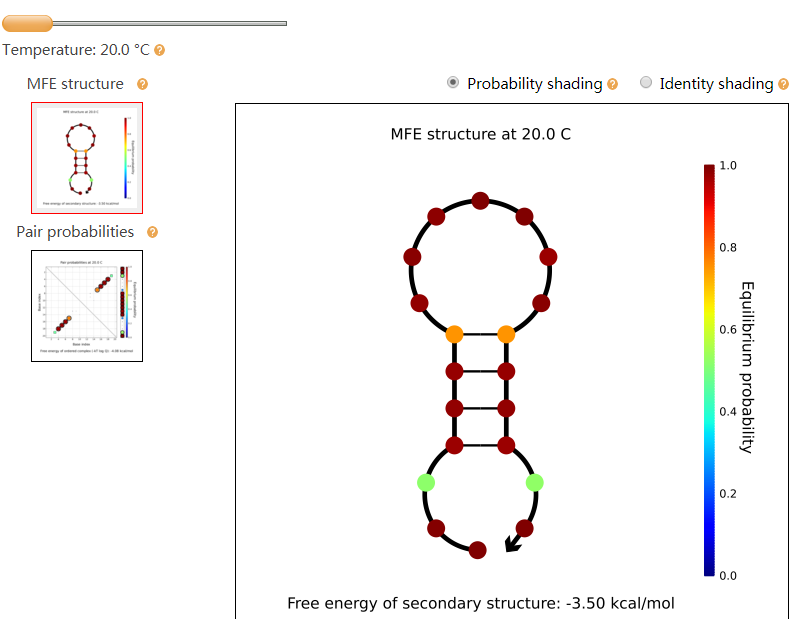
上图右侧的结构就是预测好的高级结构,图中的每一个点代表一个碱基,整个序列的3’末端用箭头表示,用以指示序列的方向性。点的颜色越接近红色,它的Equilibrium probability平衡概率也就越高,也越稳定。点的颜色越接近蓝色,这个位置就越不稳定,如果是已经形成双链的部分颜色从红色转为黄色或者绿色,说明双链更容易被解开。如果需要在图中展示具体的碱基序列,可以点击Identity shading。这时我们会看到图中所有的点都变成了四种颜色,用以代表碱基。其中绿色代表A,蓝色代表C,黑色代表G,红色代表T,如下图所示。
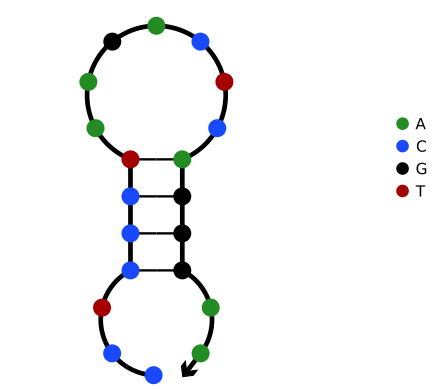
回到Probability shading的选项,图3左上角的温度那里是可以拖动的。我们把温度条从20 ℃,拖动到50 ℃,55 ℃,和60 ℃。我们会发现茎环部分的红点在50-55℃时已经从红色转变为了黄绿色,这预示着它即将解开双链,到了60 ℃时,双链部分完全解开,整条序列恢复成单链结构。
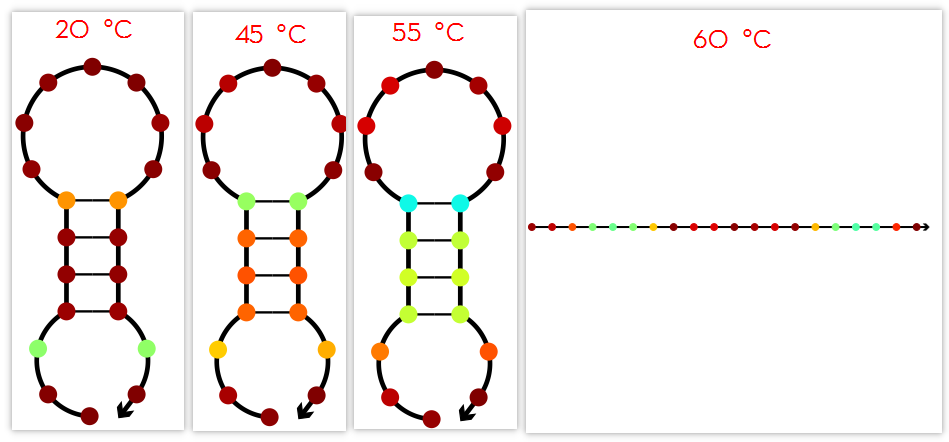
通过拖动温度条,我们可以预测到发夹结构在不同温度下的变化情况。以往我们使用其它软件得到的可能只是一个Tm值,无法直观的以结构图来进行展示。
点击左侧的Pair probabilities,可以把碱基互补配对的位置的情况图示化:
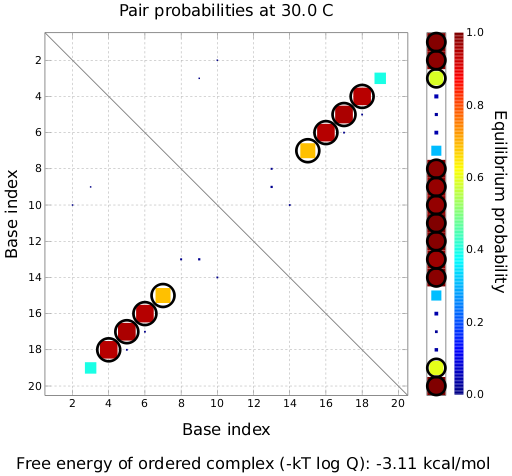
图中的横、纵坐标都是碱基的位置,从5’ 到3’。表中间被一条斜线分割成两部分,表中红黄色的点参与了碱基互补配对,如果颜色变为黄绿色,代表这个双链部分容易被解开。我们可以直观看到整条序列中的4-7,15-18部分的碱基参与了双链的形成,而图中绿色箭头所示为未配对的单链DNA位置,我们可以看到序列的两头和中间的环部位置都是未配对的。
点击Results,回到结果总览,点击Equilibrium concentrations的第二个灰色区域可以观察链与链之间形成的二级结构,分析方法和茎环结构的分析办法类似,这里不再一一叙述。
点击Histogram filters,可以进行数据筛选的设定。Contains strand species这里设置筛选核酸链的范围,如果只有一条DNA链,这里就直接选择strand 1即可。Fraction of max concentration这里设定丰度最高的二级结构所占有比例的下限,这里默认为0.001(最高为1).低于这个比例则不显示。Min concentration设置所显示的结构的浓度下限,低于浓度下限则不显示该二级结构,默认值是0. Max bars用于设置显示二级结构的数量,高于选择的数量则不显示,默认值是10.

案例2:
两条DNA链的结构分析
单链DNA的结构分析在其它很多在线网站中都可以去完成,NUPACK网站最大的优势还是可以进行多条DNA链形成的结构分析。以往我们经常会遇到这样一个问题,一条DNA链能否打开另外一条DNA链的发夹结构?使用NUPACK网站即可迎刃而解,这里我们以
H1:GTCATAGTGCAGGTAGATGGACATGGACTACCTGCACTATGAGCACTTTTAGA
和
initiator: AAAAGTGCTCATAGTGCAGGTAG为例。
在input界面将Number of strand species改为2,输入上述两条DNA序列后进行分析,其它参数都参考下图进行设置。
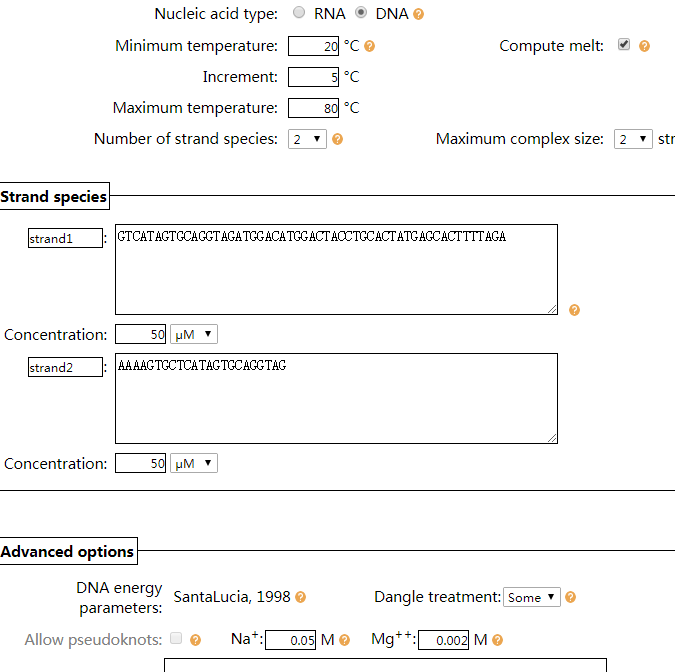
这次的结果中,在 20 ℃下只能看到strand1-strand2这一种二级结构,说明在20 ℃时strand2(initiator)能够完全打开strand1(H1)所形成的的发夹,升高温度到60 ℃时,我们注意到部分的strand1- strand2二聚体结构被打开,形成单独的strand1和strand2链。如果我们要知道恒定温度下两条DNA链结构的情况,一般为室温25 ℃或者实验温度37 ℃,这时就不用进行Compute melt的设定,只要把Temperature设定在25 ℃或37 ℃即可。应用NUPCK网站还可以预测带有错配的双链DNA的热稳定性情况,而常规软件只能预测完整的DNA双链的Tm值。
案例3:
三条DNA链的结构分析
当一条长链DNA与两条短链DNA都发生互补,形成的双链DNA中间带有缺口的情况,这种情况该怎样去预测热稳定性呢?这里我们以下面三条链为例进行预测,
A1: TACGAGTTGAGACATCCTGAATGCG,
A2:TCTCAACTCGTA,
A3:CGCATTCAGGAT。
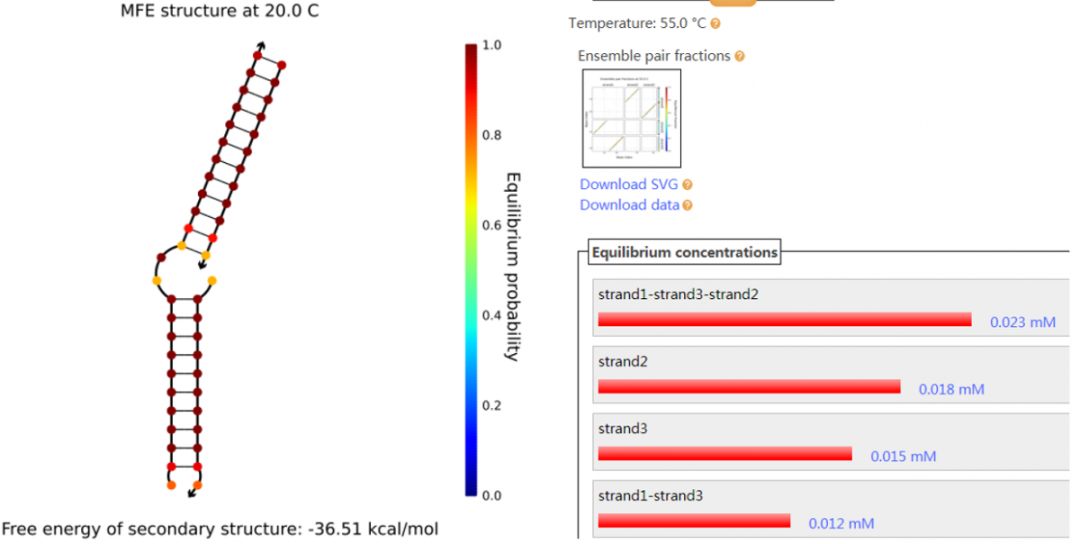
在input界面将Number of strand species改为3,分别输入三条DNA的序列后。注意Maximum complex size这里要改为3,如果设置为2的话,NUPACK就只会分析DNA两两形成的双链二级结构。其它参考图1进行操作。从图8左侧图来看,在20 ℃时,三条DNA链能形成稳定的双链,且无其它DNA结构(图中未给出)。在45 ℃时,三条DNA链形成的稳定双链开始解开(图中未给出),到达55 ℃时,已经有超过一半的双链被解开。
NUPACK软件也有其功能不足之处,就是预测的结构都是平面的。如果是四条DNA形成的正四面体三维结构,目前小编没有找到对应的预测办法。此外NUPACK软件除了分析功能,还有Design设计功能,但这就要涉及到复杂的代码编写。其功能讲解将在以后的微信文章中推出。


