断尾获新生——rhPCR
断尾获新生——rhPCR的原理、设计及应用
当壁虎、蜥蜴等动物被天敌咬住尾巴或遭遇危险的时候,往往会自断尾巴,吸引敌人的注意,乘机逃跑而得以逃生。这是一种牺牲局部,保全整体的求生方式。
而对于rhPCR来说,引物就像壁虎一样,带了一条“尾巴”,只有断尾的引物,才能重获新生,发挥扩增延伸的功能。
rhPCR 的原理
PCR的高特异性是通过引物序列的特异性来实现的,但有时为了检测目标序列,引物序列需要位于不太理想的位置。这就有可能导致引物二聚体和非特异性扩增产物的产生。
rhPCR(RNase H-dependent PCR)[1]是一套避免引物出现非特异性扩增的办法。将特异性扩增的引物靠近3’末端的位置设置一个RNA碱基,并进行3’末端的封闭。一般形式为 “rDDDDX”,其中r代表RNA碱基,DDDD代表与模板完全发生互补配对的四个DNA碱基,X代表引物3’末端的封闭性功能修饰,一般为C3 spacer。在PCR体系中加入RNase H2,酶的序列来自于热球菌目的Pyrococcus abyssi,经过密码子优化并在大肠杆菌里重组表达。Rnase H2具有热稳定性,在95 ℃孵育 90 min仍然能够保持三分之二以上的酶活性,能够适应PCR的热循环条件。Rnase H2需要一定温度才能启动它的活性,在30 ℃时,活性只有不到百分之一。在70 ℃时才能达到其70%以上的活性。这就减小了这个酶发生非特异性剪切的可能性。
rhPCR的原理是:理论上只有当特异性的引物完全与模板结合时,Rnase H2水解引物中RNA碱基与其上游核酸之间的磷酸二酯键,带有3’末端封闭修饰的RNA与DNA混合核酸序列从模板上脱离下来,暴露出带有3’末端为羟基的引物部分,这样引物才能完成延伸扩增的过程。这种对靶序列的高度互补性要求也减少了非特异性扩增的产生。
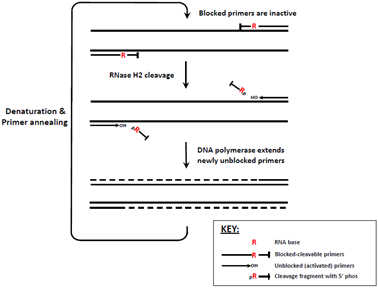
图1:rhPCR的原理
rhPCR的优点
rhPCR减少引物二聚体的产生
引物二聚体和非特异性扩增是PCR中的常见问题,在染料法荧光定量分析中尤其重要,因为从这类扩增产物会产生假阳性信号。序列分析,PCR引物的精心设计以及热启动方法的使用都可以减少引物二聚体和非特异性扩增的发生频率,但是某些情况下必须在不理想的序列范围内进行引物设计。在多重PCR扩增中,几个不同的引物对必须一起发挥良好作用的情况下,这个问题就变的更加严重。由于RNase H2在室温下的活性较低,在rhPCR中该酶的活性需要经过热启动激活。此外扩增需要引物退火并形成合适的用于Rnase H2切割的异源双链底物,与通过单独将引物与靶标杂交所提供的底物相比,rhPCR具有更大的特异性。这两者的效应合在一起,能减少引物二聚体的形成。图2是分别使用常规引物和带有RNA碱基的rhPCR引物分别扩增HCV质粒模板的结果。如所预期的,未修饰的常规引物的PCR产物出现了多个小于目标产物(242bp)的PCR产物,都是非特异性产物。这些扩增产物中,引物二聚体占据了大部分,并导致产生了低水平的HCV正确扩增产物的产生。与使用常规引物的标准PCR相比,使用可裂解引物的rhPCR引物可以产生正确的242 bp扩增产物。而当不存在HCV模板时,则没有任何扩增产物。使用可裂解的rhPCR引物可完全消除系统中引物二聚体。
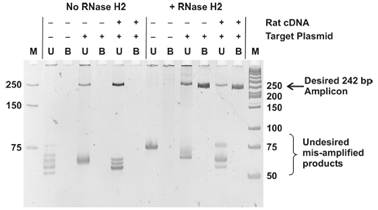
图2:rhPCR降低引物二聚体的产生
rhPCR提高了对复杂核酸样本的特异性
之前的HCV扩增结果表明rhPCR引物降低引物二聚体的产生。为了确认rhPCR引物的特异性分辨能力。针对人和大鼠同源性较高的HARS基因分别设计常规引物和带有可裂解的rhPCR引物,分别以人HeLa细胞或大鼠脊髓制备的cDNA为模板进行PCR扩增检测。如图3,以人cDNA(设计引物的真模板)为模板时,在25.9个循环观察到扩增信号,以大鼠cDNA(假模板)为模板时,在35-38个循环观察到扩增信号。相反的,通过使用“ rDDDDx”可切割的rhPCR引物,以人cDNA(设计引物的真模板)为模板时,在26.2个循环观察到扩增信号,而使用大鼠cDNA为模板时,在55个循环内未检测到扩增信号。使用90个循环再次进行扩增检测,以最大程度地检测到rhPCR引物引发错误扩增的可能性,最终在79个循环中观察到大鼠cDNA的假阳性信号。
将人HRAS对照引物和可裂解的rhPCR引物与相应大鼠Hras cDNA序列进行比对。尽管在正向和反向对照引物中都存在多个错配,但与人cDNA的扩增结果相比,大鼠cDNA的扩增结果仅存在11个循环的ΔCq延迟。rhPCR引物中由于切割位点(RNA碱基)周围存在错配,两条引物均形成了RNase H2的不稳定底物。因此rhPCR相比与对照引物相比,特异性更高。
图3:rhPCR提高了对复杂核酸样本的特异性
rhPCR的引物设计
相比于rhPCR引物的老版本“rDDDDX”,最新的rhPCR引物分为两种,它们有着不同的性质和功能。第一种版本称为“DDDDDDDDrDDDDMx”, 其中D代表DNA碱基,r代表RNA碱基,M代表错配的DNA碱基,x代表阻断基团(通常为C3 spacer)。在C3 spacer之前插入不匹配的DNA碱基以创建“ Mx”组合,可确保末端阻断基团的最大功效。第一种版本rhPCR引物适用于标准基因分型应用和多重PCR扩增。这种rhPCR引物稳定性较好,适合于和低浓度的Rnase H2配合使用。
图4:第一种版本rhPCR引物设计及原理(图片来源于https://sg.idtdna.com/pages/products/qpcr-and-pcr/custom-primers/rhpcr-primers)
第二种版本rhPCR引物为“DDDDDDDDrDxxDM”, 其中D代表DNA碱基,r代表RNA碱基,x代表封闭剂。在引物的3'末端插入错配的DNA碱基以产生“ DM”组合,从而确保了末端阻断基团的最大效力。第二种版本rhPCR引物最适合稀有等位基因检测或需要极高保真度的模板扩增应用。第二种版本rhPCR引物可能需要使用更多量的RNase H2酶(浓度是第一种版本rhPCR引物使用量的1-100倍)。第二种版本rhPCR引物需要对每一对引物的浓度和反应体系作一定调整才能正常工作,因此第一种版本rhPCR引物的适用性更广,通常情况下优先使用。
图5:第二种版本rhPCR引物设计及原理(图片来源于https://sg.idtdna.com/pages/products/qpcr-and-pcr/custom-primers/rhpcr-primers)
第二种版本rhPCR引物要避免使用rU作为RNA碱基,因为包含rU的引物比包含rC,rG或rA的引物需要更多的RNase H2酶才能有效切割。如果由于SNP分型或靶序列的限制而无法避免rU,请小心提高RNase H2的浓度,以实现rU引物的有效裂解,同时使RNase H2对反应体系中其它引物的影响降至最低;过量RNase H2的存在会降低切割的特异性。为了获得最佳性能,建议正向和反向引物都使用含有RNA碱基的可裂解的引物。但可裂解的引物与标准引物结合使用仍可比使用两条标准PCR引物获得更高的特异性。rhPCR的退火/扩展反应温度通常为60°C;但是,rhPCR在50–70°C的温度范围内都是有效的。
rhPCR的应用举例
rhPCR用于SNP检测
人与人之间的基因组序列差异有好几种,如碱基插入、缺失、SNP以及微卫星等,SNP仅是其中最常见和最普遍的一种,在临床上有很重大的意义。在使用Taqman探针法检测SNP位点时,探针遇到错配的模板,理论上是不会发生任何扩增的。但是实际上可能发生微弱的扩增,得到的CT值比探针遇到完全匹配的模板时的CT值要大得多,其中的差值为ΔCT值。使用二维的基因分型图来确认SNP位点的基因型,图6A为使用rHPCR的引物与错配模板和完全互补模板结合的ΔCT值情况,图6B为使用普通引物与错配模板和完全互补模板结合的ΔCT值情况。相比于序列3’末端与SNP位点结合的普通位点特异性引物,rhPCR引物拥有更大的ΔCT值。这些结果表明rhPCR相对于普通PCR对SNP检测有着更强的分辨能力。
图6:rhPCR引物与普通引物检测SNP时的ΔCT值情况
rhPCR用于检测真菌微生物
山地松甲虫(Dendroctonus ponderosae Hopkins;MPB)是北美西部松树的一种重要的经济和生态害虫。山松甲虫与多种微生物形成复杂的多部分关系,包括麦冬菌类。这些真菌被MPB引导到新的松树寄主,在那里真菌突破了寄主的防御能力进入树皮和树心之间。这些病变代表一种感染症状,代表真菌生长和树木防御能力。为了更客观地评估真菌的毒力和寄主-病原相互作用研究中的寄主树敏感性,可以使用比普通PCR特异性和敏感性更高的rh-qPC方法。G. clavigera是MPB中最常见的真菌共生物种,也被认为是最具致病性的。图7A使用28 S通用引物,针对所检测到的麦冬属真菌中的一个rDNA区。图7B使用Gc引物,针对β-微管蛋白,设计用于区分G. clavigera和其它与MPB相关的麦冬属真菌。结果表明,28 S引物可以稳定性的检测到9种麦冬属真菌。Gc引物能有效的检测到G. clavigera,对其它几种麦冬属真菌几乎没有扩增或扩增性较弱[2]。
图7:rhPCR引物用于检测麦冬属真菌
“
参考文献
1. Dobosy JR, Rose SD, Beltz KR, Rupp SM, Powers KM, Behlke MA, Walder JA, RNase H-dependent PCR (rhPCR): improved specificity and single nucleotide polymorphism detection using blocked cleavable primers, BMC Biotechnol. 2011 Aug 10;11:80. doi: 10.1186/1472-6750-11-80.
2. McAllister CH, Fortier CE, St Onge KR, Sacchi BM, Nawrot MJ, Locke T, Cooke JEK. A novel application of RNase H2-dependent quantitative PCR for detection and quantification of Grosmannia clavigera, a mountain pine beetle fungal symbiont, in environmental samples. Tree Physiol. 2018 Mar 1;38(3):485-501.

