您是否遇也到过这种情况:转录组和蛋白质组相关性比预期的要低,经历千辛万苦好不容易从海量的转录组数据中筛到了一些与研究方向最相关的显著差异表达基因,但通过蛋白质组检测的时候确发现对应的生命执行者蛋白质变化并不显著甚至变化相反,直接做转录组+蛋白质组貌似并没有那么顺畅的获得想要的信息,究竟是什么原因呢?
原来转录组和蛋白质组相关性差是翻译调控搞的鬼【1】,目前对翻译调控的研究又被称作“翻译组学”。
那么用于研究翻译组的主流技术方法有哪些呢,为什么单单Ribo-seq 被称为解码翻译组学的“黑匣子”破译工具?我们带着这些问题一起来揭开前言技术Ribo-seq的面纱吧。
Ribo-seq(Ribosome Profiling,也称翻译组测序,核糖体印记测序)是⼀种通过对与核糖体结合的正在翻译的RNA片段进行⾼通量测序,来研究翻译过程中的核糖体⾜迹(包括mRNA和其他潜在的可翻译RNA分子如lncRNA、circRNA等)的技术。Ribo-seq能够提供关于翻译效率、核糖体占有率和蛋⽩质合成动态的详细信息,是连接转录组与蛋白质组之间的桥梁。
由于核糖体对与之结合的~30nt mRNA序列具有保护作用,利用RNA酶消化细胞中的RNA后,这些被核糖体保护的RNA片段可以被富集,通过对这些片段检测和分析不仅能获得全面的、高质量的蛋白质翻译速度情况,了解蛋白质表达水平及其丰度,还能直接对翻译过程进行研究。而结合转录组、小RNA测序、蛋白组测序等进行关联分析,可以更精确地研究转录后调控、翻译调控机制。
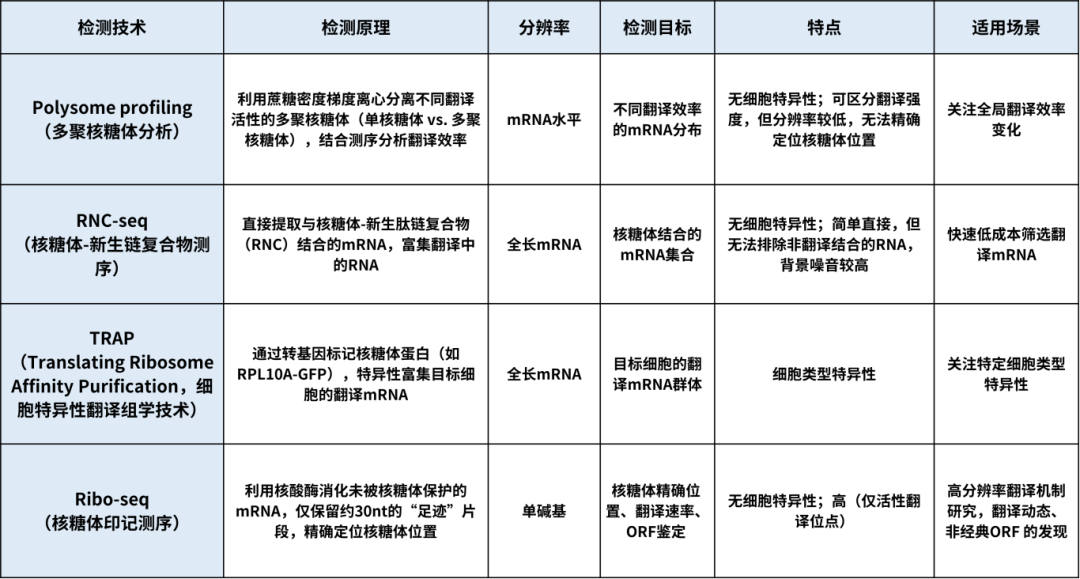
因此我们可以根据关注点进行翻译组学技术的选择:
关注全局翻译效率变化? → 选 Polysome Profiling
快速低成本筛选翻译mRNA? → 选 RNC-seq
需要细胞类型特异性? → 选 TRAP
需单碱基精度的翻译位点? → 选 Ribo-seq
ü 单碱基分辨率,精准识别翻译起始位点(TIS)和开放阅读框(ORF)
ü 是唯一能解析核糖体占据位点的技术,上述其它方法均无法区分mRNA是否正在被翻译或仅与核糖体结合
ü 区分翻译与非翻译 RNA,降低噪音
ü 发现非经典翻译事件(如 uORF、微小肽等)
-
对翻译调控机制的研究(如应激响应、疾病发生)
-
对新蛋白、微小肽的挖掘
-
深化转录组测序分析
-
解释转录组和蛋白组结果不一致的原因(可能因为基因转录而不翻译或者翻译效率低;而蛋白组仅能粗略定位到基因区域,无法明确转录本及编码的ORF)

注:除模式动物外,其他物种需提前咨询
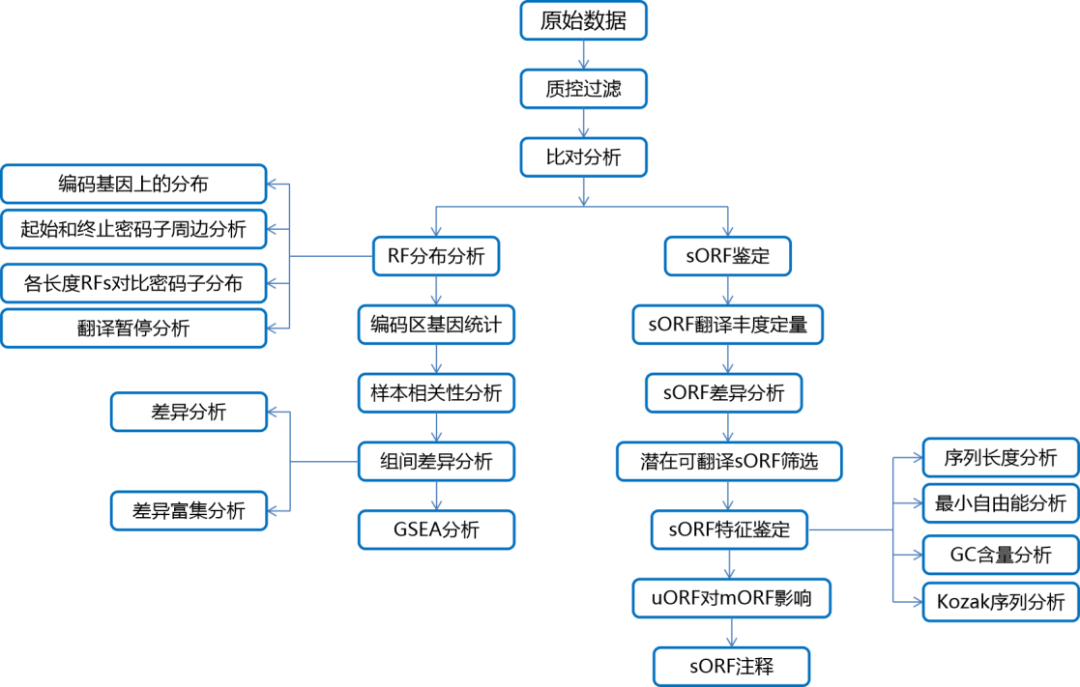
Ribo-seq 如同为翻译过程安装了“分子级监控”,使研究者可以“看见”核糖体的每一步移动,量化翻译速率与效率,同时可以发现隐藏的遗传信息,将蛋白质合成的不可见动态转化为可解码的数字信号。因此,Ribo-seq被形象喻为解码翻译组学,揭示蛋白质合成的“黑匣子” 。另外,有统计称转录组与蛋白组的相关系数通常仅0.4-0.7(酵母至人类均如此),而翻译组填补了这一信息断层,将断裂的基因表达信息串联为完整故事链,真正实现了从序列到功能的闭环解析。翻译组学聚焦于 mRNA 的翻译动态,揭示哪些 mRNA 正在被翻译、翻译效率如何,以及翻译调控机制。相比转录组(mRNA 水平),翻译组更直接反映蛋白质合成的真实状态,是连接基因组与蛋白质组的桥梁。转录组,蛋白组,翻译组三者处于基因表达的不同层级,动态范围和调控机制各异,通过联合分析转录组(潜在能力)、翻译组(执行选择)和蛋白组(最终效果),可更精准地解码复杂生物学系统。
如果老师对转录组,蛋白组,翻译组感兴趣,尽快联系我们吧,做多组学项目的话,还可以赠送联合分析吆~~
技术支持
电话:021-57072059/2097/2177/2179
邮箱:RNAseq@sangon.com/RNAseq2@sangon.com/RNAseq3@sangon.com
参考文献:
【1】Schwanhausser B, Busse D, Li N, et al. Global quantifcation of mammalian gene expression control. Nature, 2011, 473(7347): 337-342.

