SNP (Single Nucleotide Polymorphisms)全称单核苷酸多态性,是指在基因组上单个核苷酸的变异,变异类型包括转换、颠换、缺失和插入。SNP是人类可遗传的变异中最常见的一种。占所有已知多态性的90%以上。SNP在人类基因组中广泛存在,平均每500~1000个碱基对中就有1个,估计其总数可达300万个甚至更多。作为第三代遗传标志,人体许多表型差异、对药物或疾病的易感性等等都可能与SNP有关。因此,对SNP的检测具有重要的生物学及医学意义。
Taqman探针法将样品与反应组分在封闭的体系中进行简单混合,通过检测PCR反应过程中的荧光变化,可以实时检测SNP位点的基因分型。由于无需任何PCR之后的操作,这样就避免了PCR之后的人力成本与PCR后处理过程中的交叉污染。由于PCR反应的灵敏性,这种方法需要的样本含量极少。同时,随着日益成熟的荧光定量PCR仪,可以在一个反应板中进行大批量样本的同时检测。借助于以上优点,Taqman探针法荧光定量PCR检测成为最常见的SNP分型方法之一,并在如火如荼的分子诊断领域大放异彩。
无论是否用于SNP分型,Taqman探针的检测原理是一致的,即利用Taq DNA聚合酶的5-3核酸外切酶活力对探针本身进行酶切 (注意,pfu类型的酶缺乏5-3核酸外切酶的活性,不能用于Taqman探针法)。Taqman探针的两端分别标记有荧光基团和对应的淬灭基团。当探针完整时,oligo相当于一条锁链将荧光基团和淬灭基团紧紧拴在一起,导致荧光基团发出的光被淬灭基团吸收(FRET)。当探针被Taq酶切断之后(事实上其他的核酸酶也可以切割,如DNase I),荧光基团得以释放从而导致荧光值上升。荧光值的变化会被荧光定量PCR仪或者其他检测仪器记录下来。
下图显示了荧光探针如何借助5’核酸外切酶活性来进行等位基因分型。

对于SNP分型检测,体系中会含有针对不同基因型的探针(如下图Probe1和Probe2),两种探针5端通过标记不同的荧光基团进行区分(通常使用的是FAM和VIC)。他们的序列中包含匹配不同基因型的碱基,碱基错配将导致探针与模板的结合能力以及被切割的概率大大降低。因此,当基因型为纯合子时,只会检测到单独一种荧光信号,而对于杂合子,两种荧光信号都将被检测到。
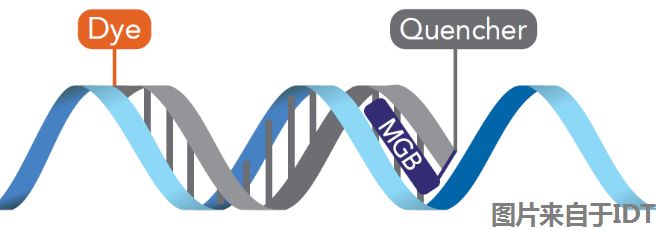
我们知道,当荧光基团和淬灭基团距离靠近时会得到比较好的淬灭效果,然而通常情况下,为了简化探针的设计与合成难度,我们会将荧光基团和对应的淬灭基团分别修饰在探针的5'端和3'端(早期也有反过来修饰的)。虽然这样的修饰位置使得荧光、淬灭基团距离较远,看起来与常理违背,但实际上,游离的单链探针在溶液中是扭曲状态,也能达到比较好的淬灭效果。
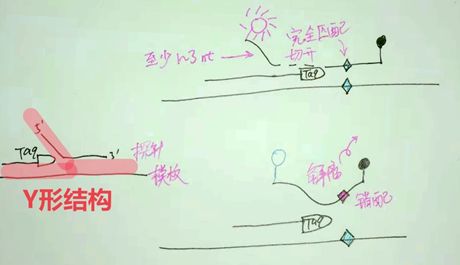
有人为了增加淬灭效率,将荧光基团、淬灭基团修饰在临近的位置:如5端修饰FAM、第3个碱基上修饰BHQ;或者倒数第3个碱基修饰FAM、3端修饰BHQ。以上都是不推荐的。当这两个基团都在5端时,探针的切割可能发生在它们的下游;当这两个基团都在3端时,有可能探针提前脱离模板,这些情况都会导致切割失败,信号丢失。如下图:

影响探针对SNP分型效果的因素有很多。
首先,探针中错配的碱基需要对整条探针与模板的结合能力有比较大的影响,即含有错配的探针Tm值需比正常探针低得多。这对探针的长度及SNP位点在探针中的位置有较高的要求。
第二,由于分型探针是在同一个反应体系中,因此不同的探针与模板的结合是一种竞争状态,这就要求不同的分型探针与模板结合的位置是需要重叠的,这样才能保证一个模板上只结合一种探针。
第三,Taqman探针的水解是先解离一部分,然后再水解。Taq酶的5’核酸外切酶活性实际上识别的是一个Y型结构,其游离的单链至少为1~3个核苷酸,此时要求错配的探针应当更倾向于从模板上解离下来,而不是继续留在模板上被Taq酶水解。
上文说到,SNP分型探针在发生碱基错配(如野生型探针结合到突变型模板)时,需要有比较大的热力学差异,导致Tm值降低。探针长度越长,单个碱基错配对整体探针Tm影响越小,野生型和突变型探针也就越类似,这导致了探针分型灵敏度的下降,容易产生误判。因此,长度短的探针区分能力更强。
Epoch的科学家发现,当Oligo绑定上一段小沟结合物(MGB)之后,其与模板的结合稳定性大大增加,结合在探针3端淬灭基团旁边时,也能够实现比较理想的分型能力。由于Tm值的大幅提升,MGB的加入缩短了分型探针的长度,更适合进行单核苷酸的分型。
下方列出了SNP分型探针的设计原则及优先度,在不能全部满足下方的要求时,尽量满足优先度靠前的原则。
1. 避免5’端第一个核苷酸为G,鸟嘌呤核苷酸对临近的荧光基团有明显的淬灭作用,导致结果信号值低。
2. 探针的Tm值需要比引物的Tm值高5~8°C,通常情况下为65~67°C
3. 为了增加探针的单碱基区分度,探针长度不宜过长,但同时MGB探针也不宜短于13个核苷酸。
4. 避免连续相同碱基的出现,尤其是连续的G,多余4个连续的G存在时,探针容易形成G四联体或其他高级结构,不利于其与模板的结合。
5. 区分SNP的碱基应当位于探针中间1/3的位置去(将探针平分三段,SNP位于中间那段)。如果这段位置不合适,设计不出优秀的探针,可以将SNP位置向3’端挪动,以更加靠近MGB基团。不要将SNP分型位点安排到最后的三个核苷酸中。
6. 由于G核苷酸形成高级结构及对荧光团的淬灭特性,探针的G残基数量不宜超过C的数量,如果有此情况,建议将探针设计在互补链上。
小编最常使用的软件为ABI公司的Primer Express 3.0,支持XP系统及Win7系统兼容模式,Win10小编如何打开都不行,关于该软件的使用,小编将在后续的推送中分享给大家。另外,PrimerBiosoft公司的AlleleID软件也可以进行SNP分型探针的设计,有兴趣的小伙伴可以尝试一下


