1.1 登录NCBI官网,https://www.ncbi.nlm.nih.gov/
1.2 点击All Resources
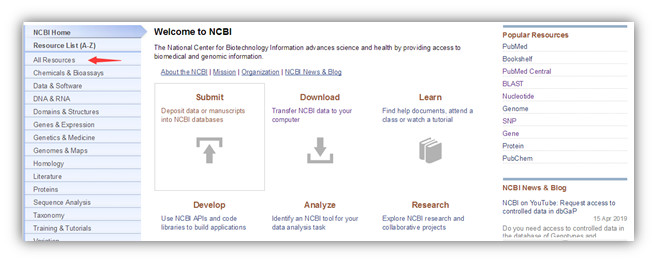
1.3 再点击Downloads,之后点击BLAST (Stand-alone),
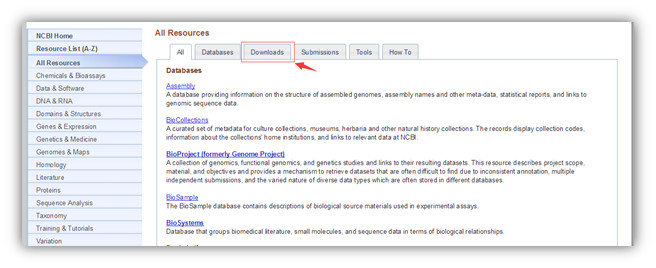
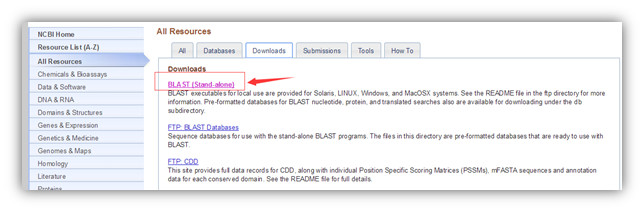
1.4 找到下载链接并点击下载
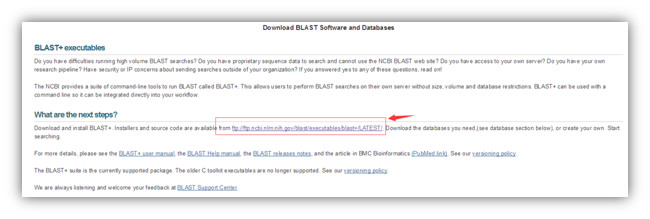
1.5 下载适用版本的可执行文件(我们自己的示例电脑为64位操作系统的,因此下载64位的)
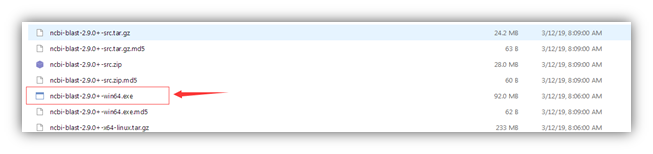
1.6 下载完成后,默认安装到本地电脑
2.1 适用win+R快捷键,然后输入cmd(或者点击开始-运行-输入cmd),进入DOS命令界面
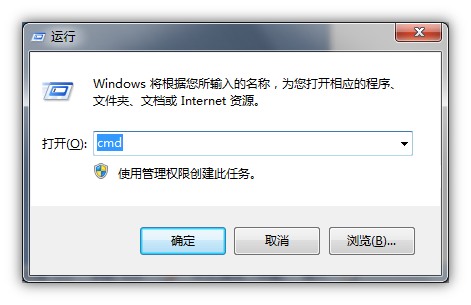
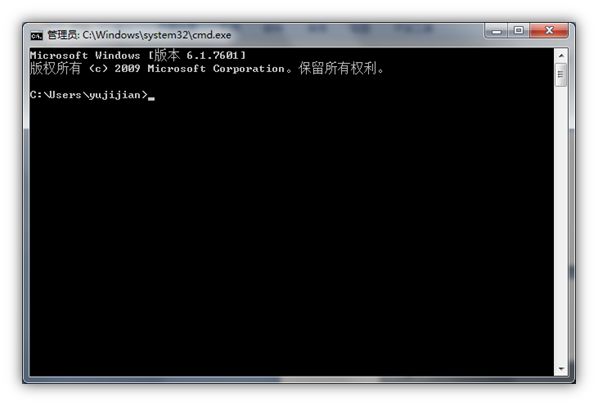
2.2 创建比对工作的文件夹
输入命令:md localblast

注意md后面有个空格;
输入命令:dir查看创建成功与否

也可以在C盘内看到该文件夹
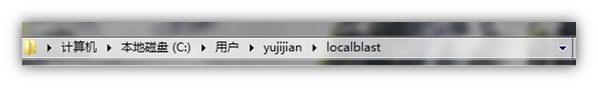
2.3 准备比对文件
分别准备需要比对的基因序列文件(fasta/fa格式)和被比对的全部转录本文件,也就是数据库来源,亦为fasta/fa格式。
备注:fasta格式文件为行以“>”开头记录序列id,然后另起一行记录序列碱基组成,如下图所示:

文件准备好后,如下:
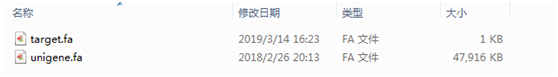
2.4 构建数据库
先进入目标文件夹,输入命令:cd localblast
然后输入如下命令:makeblastdb -in input_file -dbtype nucl -out database_name
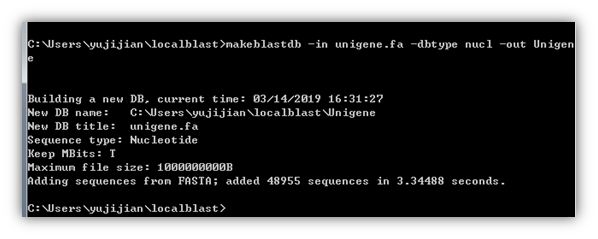
文件夹中会多出一些文件,这些文件即是数据库文件,如下:

input_file:数据库的文件名称,例如unigene.fa
nucl:数据库的性质,本文中nucl为核酸序列,也可以选择prot,为蛋白序列
database_name:数据库名称,如不填,则为输入文件名称
其它更多参数信息,可以输入命令makeblastdb -help进行查看。
备注:每个字段间必须使用空格隔开。
3.1 输入DOS命令:blastn -query target.fa -db Unigen -out blastout.txt -evalue 1e-5
blastn:调用的比对软件模块,该模块为核酸序列比对核酸序列。也可以调用blastx执行核酸序列比对蛋白序列功能。
-query target.fa:需要比对的序列文件
-db Unigene:数据库
-out blastout.txt:比对结果输出文件
-evalue 1e-5:比对期望值,默认设置为1e-5
3.2 在工作目录下,打开blastout.txt文件,查看比对结果。

