密码子优化,没~那么简单~
近日小编收到一个师妹的咨询,已知一段氨基酸序列,如何才能逆向翻译成为DNA序列,愉快地在哺乳动物细胞中表达呢?
活细胞将DNA和mRNA中编码的遗传信息转化为蛋白质。该编码信息由三联核苷酸组成,称为密码子。通常情况下,总共有64种不同的密码子。其中的61种编码20种标准氨基酸,另3种起终止密码子的作用。相对于氨基酸种类而言,密码子的种类更多,这意味着一个氨基酸可以由多个密码子编码。事实上,一些常见的氨基酸,如精氨酸和亮氨酸,由多达6个密码子编码。
对于相同的氨基酸,不同的生物表现出对某些密码子使用的偏好性。有些物种几乎完全避免了某些密码子的使用。因此,在进行表达研究时考虑密码子优化是很重要的。如果密码子选取不当,会造成表达量降低、表达缓慢、提前终止等后果。
密码子优化是什么感觉?读下面的文字就体会到了。

因此,我们必须根据表达宿主的密码子偏好性,将我们的序列进行优化,让宿主不仅能读得懂,并且读的通。
根据物种密码子偏好性?
没这么简单
以人源细胞宿主为例,下图是Homo Sapiens 的密码子偏好性表(来自于SnapGene),最常用的密码子以绿色背景显示。
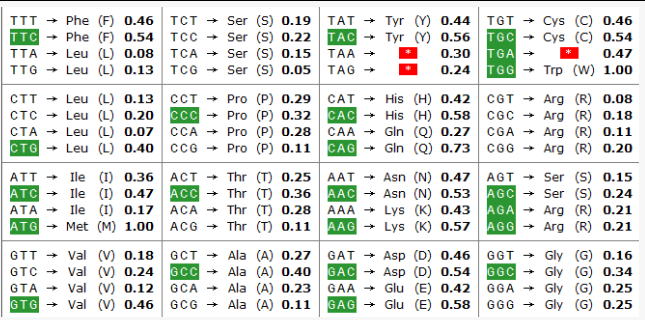
表1:Homo Sapiens 的密码子偏好性表
那么是不是将DNA序列中的密码子比例换成上述这些比例就可以了?
不是这么简单~
下图是小编随机选取了一段氨基酸序列,使用不同的方法进行优化、逆向翻译,之后与上图的偏好性进行对比:
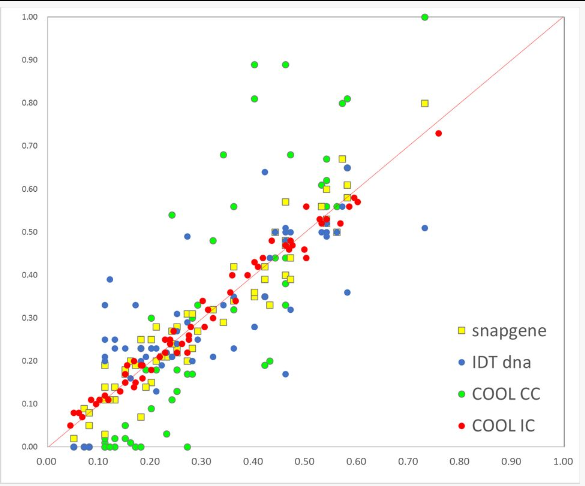
图1:使用不同方法优化的偏好性散点图
上图中每一个散点都对应一种密码子,越靠近红色斜线,说明其与标准密码子偏好性越符合;越离散,说明其与标准的比例差异越大。
可以看出,snapgene与COOL IC两种方法(■、●)非常聚集,所以是按照表1中的密码子偏好性来进行优化的,下面图片是COOL IC与表1中的偏好性对比,可以说是几乎一致的。
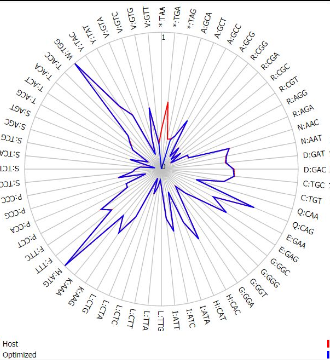
那么是否说明其他两个方法(IDT/COOL CC)优化的不好?并不是,只能说明,snapgene与COOL IC两种方法使用的仅是常见的优化标准之一,并不是唯一的优化算法。
这里小编列举几种常见的密码子优化算法:
1. Individual Codon Usage (ICU) :独立密码子使用,指输出的DNA序列与表达宿主的密码子使用频率匹配。snapgene与COOL IC使用的就是该种方法。
2. Codon Context (CC):密码子前后关系,类似于ICU,但与宿主匹配的是相邻两个密码子(总共有6个核苷酸),而不是每个单独的密码子。例如,对于YW这两个相邻的氨基酸,在人源宿主内的密码子有两种:TACTGG(占比75.16%)和TATTGG(占比24.84%)。那么在优化之后的YW氨基酸的DNA序列将尽量按照上述比例。
3. Codon Adaptation Index (CAI):密码子适应指数,是指同义密码子与最佳密码子使用频率的相符程度,取值在0~1之间。以高表达基因的序列为参考,评估目的基因与参考序列的密码子使用频率相符程度。例如,一个宿主使用苯丙氨酸的密码子分别为TTT(20%)和TTC(80%),当有一个TTC出现时,打分=0.8/0.8=1;而当有一个TTT出现时,则打分=0.2/0.8=0.25。所以,当以CAI为主要指标进行优化时,序列将尽可能使用最偏好密码子。
综上,
-
Snapgene与COOL IC使用的是ICU方法。
-
COOL CC,则是用的Codon Context的优化参数。
-
IDT则使用了一套自己的方案:将低于10%偏好性的密码子去除,重新分配剩余密码子的偏好性,然后根据ICU算法进行优化。
我们很难说哪种优化方案是最好的,目前尚没有定论。但至少按照上述一种或多种算法组合,我们可以顺利的将氨基酸序列,根据表达宿主的物种,进行逆向翻译成为DNA序列了。
密码子优化绝不仅仅是对密码子序列的调整和改变,由于密码子优化之后往往用于蛋白的表达,涉及到从载体构建、转化、RNA转录到蛋白翻译的所有过程。因此,必要时,还需要考虑以下几个影响因素:
CpG位点:主要涉及到DNA的甲基化影响
GC含量:影响 mRNA 热力学稳定性及二级结构
重复序列:影响DNA合成的难易度以及转录RNA的高级结构,RNA的5端如果形成高级结构会阻碍翻译过程的顺利进行
特定的Motif:密码子优化有时候可能会破坏某些原本存在的DNA motif,在优化时需要避开其区域
酶切位点:如果在后续构建载体过程中需要用到某些酶切位点,需要在优化时避开其序列
其他因素:如终止密码子“伏击”假说(ambush hypothesis)、mRNA自由能等等
好了,密码子优化方面今天就说这么多,能看到这里的,小编给你们点赞!
如果大家有密码子优化或者基因合成的需求,欢迎大家咨询生工生物基因中心:
邮箱: gene@sangon.com
祝您十一长假快乐!

.png)
