1. 基因组重测序
2. SNP和SNV的区别
3. 基因组覆盖度和基因组测序深度区别
4. 共有突变和差异突变
5. 基因组重测序能分析多大的插入缺失突变
基因组重测序
基因组重测序是指,对一个有参考基因组的物种的不同个体,进行基因组测序。而后将测序所得的数据和参考基因组进行比对分析,查找突变信息的一种测序项目。
根据上述定义,可以知道其基本特征:
A) 必须有参考基因组;
B) 必须是同一个物种的参考基因组,对细菌而言,必须是同一个菌株的参考基因组;
C) 分析策略是不同个体相对参考基因组的突变;
D)该分析策略不做基因组拼接,因此实际不会获得被测序样品的基因序列。
所以,如果没有参考基因组的物种,拿近缘物种参考基因组来比对,严格意义上说,是不可以的。因为两个物种间基因组肯定会有一些基因序列是不一样的。另外,对于细菌而言,最好是同一个菌株是有参考基因组的,同一个物种,但是不同菌株的细菌,基因组也有一些差别。
SNP和SNV的区别
SNV, 是single nucleotide variant的缩写,其定义是单碱基突变。合理的解释是,在单个个体上,其基因组上某个碱基被其它碱基替换了。而SNP则是single nucleotide polymorphism的缩写,其定义是群体中,突变率大于1%的单碱基多态性,是可遗传的突变。
以人基因组为例,假如,某个正常人,抽一管血,去检测rs2043556(T>C)位点的基因型,那么他只有三种可能,T/T纯合子,T/C杂合子以及C/C纯合子三种情况,而且这个位点是可遗传的。这就是我们说的SNP,即单碱基多态性,携带突变的人不一定患病。
但是,假如,某个人不幸罹患肿瘤,医生手术切割其肿瘤组织(不免会切下正常细胞),将这份样品拿去测序,可能肿瘤细胞内就有一些单碱基突变。这个时候,对这份样品而言,其突变率可能只有10%,因为还有其它正常细胞没有突变。而且,这种肿瘤细胞的突变是不会遗传给后代的。这种单碱基突变就称为SNV。
在实际分析数据中,经常搞混SNV和SNP。其实SNV是SNP的来源,而且对单个样品而言有许多SNV本身也是和SNP重叠的。
基因组覆盖度和基因组测序深度区别
在细菌基因组重测序中,测序深度(偶尔也有称为覆盖度)经常表述为sequencing depth,更严格的说,是平均测序深度。其计算公式是:测序深度=(总测序序列条数×序列平均长度)/基因组大小。大约意思就是,基因组上平均每个碱基被测到了几次。实际情况是,由于基因组不同区域GC含量等不同,实际不同区域的测序深度还是有区别的。
而覆盖度则表述为breadth of coverage,含义是测到的有效序碱基数列占参考基因组大小的百分比,这个比值自然是越大越好,这样没有被测到的区域越小。外显子捕获测序中,这个覆盖度意思是中靶率。
共有突变和差异突变
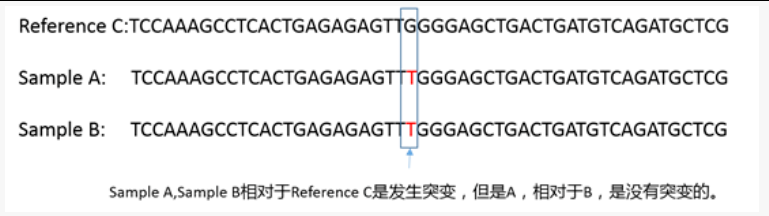
首先,要理解,相对于参考基因组而言,它们都是突变。其次,突变都是相对而言的。举个例子,如果做了2个样品,编号分别是A、B,它们分别和参考基因组C比对分析突变情况。如果某个位置,A、B两个样品都突变了,由G突变为T,它们二者相对于序列C是突变了,但是A和B相对而言,是没有突变的。
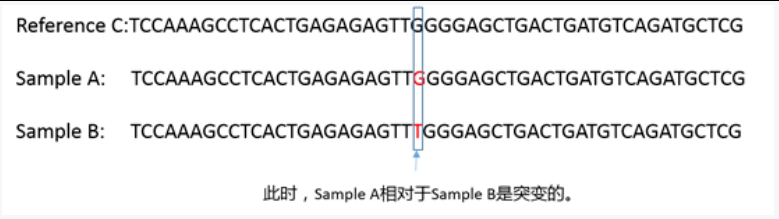
所以,当做很多样品基因组重测序的时候,需要对样品分组,如果是要找样品间差异突变的,还需要搞清楚差异是谁相对谁而言的。有的时候,A相对于B和B相对于A发生的突变是不一样的。
基因组重测序能分析多大的插入缺失突变?
一般只能分析50bp以内的插入缺失突变。主要是因为,主流的建库插入片段长度都在200-300bp,测序模式都是双端各测150bp。如果插入/缺失片段大于50bp以上,在单个序列上无法直观体现出来。很多区域也不能确定是没有测到还是真的缺失。
.png)

