通常我们认为DNA含有四种核苷酸,分别是腺嘌呤、胸腺嘧啶、鸟嘌呤和胞嘧啶,在许多生物中,胞嘧啶是几种共价修饰的靶点,如甲基化胞嘧啶(5-methylated cytosine,5mC)、羟甲基化胞嘧啶(5-hydroxymethylcytosine,5hmC)等[1]。
5mC是在 DNA 甲基化转移酶(DNA methytransferase,DNMT)的作用下,在基因 DNA 序列 CpG 二核苷酸(CpG dinucleotides,基因序列中5’→3’方向紧随相连的CG碱基,即5'-CG-3’)的胞嘧啶5’-碳位共价键结合一个甲基基团(methyl group,-CH3)而形成 5mC [2]。当然胞嘧啶除了5mC这一种变体之外还会产生一系列其他变体,其产生的过程如下图1所示。
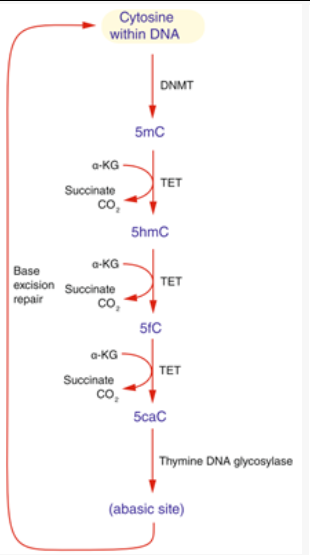
图1 胞嘧啶变体及其产生[3]
DNA甲基化与许多细胞过程、X染色体失活、胚胎发育、染色质结构的改变密切相关[4,5]。全基因组DNA甲基化状态分析表明,DNA甲基化在不同类型的肿瘤、肿瘤的各个发展阶段均有差异。研究全基因组DNA甲基化图谱可反应基因型与表型关系,为评估肿瘤风险和预后提供指标,从而为癌症的预防、诊断和治疗提供一个新途径[6]。
图2展示了几种常用的全基因组甲基化的研究方法,其中a部分是将基因组DNA片段化,基因组DNA可以被MBD富集、抗体富集、亚硫酸氢盐转化或TET氧化这几种方式来进行研究,后续通过微矩阵或者高通量平台进行分析。B部分展示的是单细胞DNA甲基化分析。
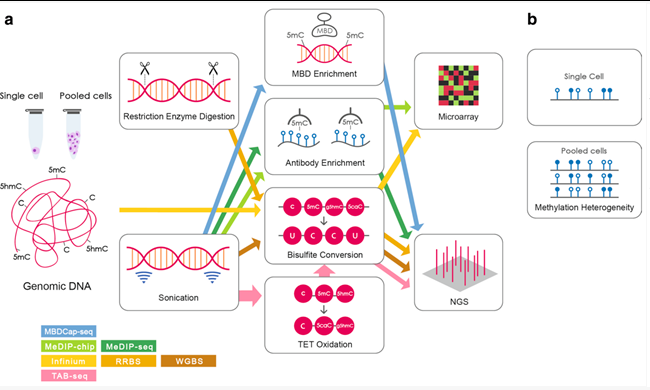
图2 常用的全基因组甲基化的研究方法[7]
我们项目服务使用的研究方式是全基因组 DNA 甲基化测序(Whole Genome Bisulfite Sequencing, WGBS)。WGBS被视为 DNA 甲基化研究的“金标准”,它通过利用重亚硫酸盐处理 DNA导致未甲基化的C变成U并在后续PCR和测序过程中成为T,而甲基化的C不受重亚硫酸盐的影响,后续对处理后的基因组DNA建库测序,可对整个基因组上的甲基化情况进行分析,具有单碱基的分辨率,可精确评估单个C碱基的甲基化水平,覆盖范围广。它可以构建精细甲基化图谱,建立表观遗传学研究数据库,为后续大规模开展不同样品间的甲基化差异分析提供参考图谱。
生工生物工程(上海)股份有限公司自2011年成立高通量测序部以来,逐步引进了Illumina,Life,DNBSEQ,PacBio、Nanopore、等二代/三代测序平台。建立了2000平米高标准实验室(通过了国家ISO认证)并搭建了50PB级存储及运算能力的服务器集群,能满足绝大多数科研客户的研究需要。同时,生工高通量测序团队建设也在不断完善中,目前有100余专职员工,其中高级技术人员以及高级生物信息工程师占比50%以上,10年来,通过生工高通量测序服务所发表的文章累计影响因子在10000分以上。
我们的新服务全基因组甲基化测序也即将全新上市,欢迎感兴趣的老师来电咨询。
技术支持电话:021-57072057/2096。
参考文献
1. Smith ZD, Meissner A. DNA methylation: roles in mammalian development. Nat Rev Genet. 2013;14:204–20.
2.Bestor TH.The DNA methyltransferases of mammals.Hum Mol Genet,2000,9(16):2395-402.
3. Netha Ulahannan and John M Greally. Genome-wide assays that identify and quantify modified cytosines in human disease studies. Epigenetics & Chromatin, 2015,8(5).
4.Wilson GA, Dhami P, Feber A, Cortázar D, Suzuki Y, Schulz R, et al. Resources for methylome analysis suitable for gene knockout studies of potential epigenome modifiers. Gigascience. 2012;1:3.
5.Jones PA. Functions of DNA methylation: islands, start sites, gene bodies and beyond. Nat Rev Genet. 2012;13:484–92.
6.张晓云. 全基因组甲基化研究进展,2013,29(10):637-639.
7.Wai‑Shin Yong,Fei‑Man Hsu,Pao‑Yang Chen. Profiling genome-wide DNA methylation, Epigenetics & Chromatin (2016) 9:26.

