Cas蛋白的体外应用
CRISPR全称Clustered Regularly Interspaced Short Palindromic Repeats,成簇的规则间隔短回文重复序列,Cas(CRISPR-associated)是CRISPR相关蛋白的简称,CRISPR与Cas蛋白组成CRISPR-Cas系统。该系统的主要作用原理是成熟的crRNA与tracrRNA配对形成单向导RNA(sgRNA),从而引导Cas蛋白在特定的核苷酸靶位点处切割双链DNA,由此引发2种不同的DNA修复机制,即非同源重组(non-homologous end joining, NHEJ)和同源重组(homology-directed repair, HDR),从而实现对基因的编辑。CRISPR-Cas系统最初来源于在原核生物中发现的细菌自身抵御入侵病毒的自然免疫过程,2013年,研究人员证明可将CRISPR-Cas依赖性基因组编辑用于哺乳动物细胞,自此CRISPR-Cas基因编辑得到了快速的发展和应用,CRISPR-Cas系统的数量和多样性也在近年来大幅度增加。
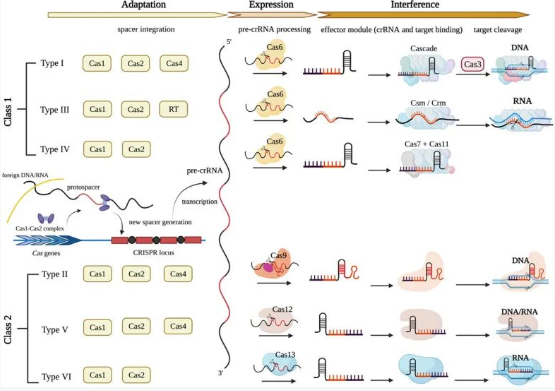
Fig.1 CRISPR-Cas系统在细菌中的分类和功能[2]
CRISPR和CRISPR 相关(Cas)蛋白系统已经改变了基因组编辑和转录调控领域。CRISPR-Cas 技术的进展也促进了从核酸到蛋白质等多种靶标的分子检测。将 CRISPR-Cas 系统与各种核酸扩增策略结合使得能够产生扩增的检测信号,富集低丰度分子靶标,提高分析特异性和灵敏度,以及开发护理点(POC)诊断技术。这些系统利用各种 Cas 蛋白的特殊特征,包括 RNA 引导的内切核酸酶活性,序列特异性识别,Cas12和 Cas13的多次翻转反式切割活性以及 Cas9的解旋和切割能力。在核酸扩增后整合 CRISPR-Cas 系统,由于 RNA 引导识别扩增子的特定序列,提高了检测特异性。在核酸扩增之前加入 CRISPR-Cas 能够富集稀有和低丰度的核酸靶标并消耗不需要的丰富核酸。在中等温度下使用 CRISPR-Cas9将 dsDNA 展开成 ssDNA 有助于实现核酸的等温指数扩增技术。CRISPR-Cas 系统与功能性核酸(FNA)和分子翻译器的组合使得能够检测非核酸靶标,如蛋白质,金属离子和小分子。成功地将 CRISPR 技术与核酸扩增技术相结合,可以高度灵敏和快速地检测出的SARS-CoV-2病毒。
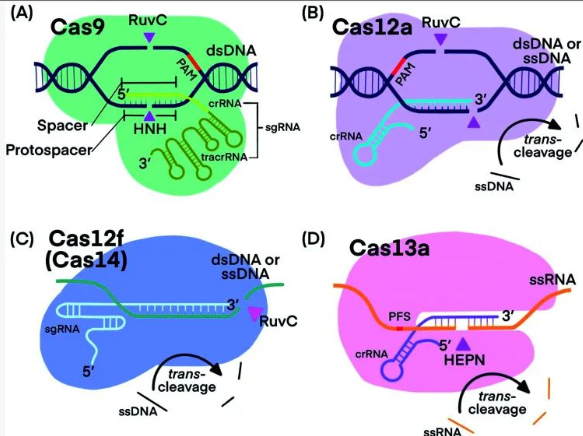
Fig. 2 CRISPR–Cas9, -Cas12a, -Cas12f, and -Cas13a复合物结构[3]
生工推荐
生工生物新推出了Cas蛋白系列产品,当前主要包括Cas12和Cas13,可用于CRISPR诊断相关实验需求。这里以重组 LbCas12a (Cpf1)为例:
重组 LbCas12a (Cpf1)是通过携带编码来自Lachnospiraceae bacterium ND2006 菌株的LbCas12a (Cpf1)基因的质粒导入大肠杆菌之后表达纯化而来,是一种RNA引导的核酸内切酶。重组 LbCas12a (Cpf1)可依托于 PAM序列特异性地剪切靶标双链DNA生成粘性末端,也可以不依赖PAM 序列特异性剪切单链靶标DNA(顺式剪切)。此外,双链或单链DNA 靶标均能激活 LbCas12a 的反式剪切活性(即旁路剪切活性/附属剪切活性),即当 LbCas12a 酶与 crRNA、靶标 DNA 结合形成三元复合物后,便会被激活针对非特异序列 ssDNA 的反式剪切活性,将体系中的任意序列ssDNA切碎。因此,LbCas12a 酶不仅可用于体外 dsDNA 的特异剪切,也可用于靶标核酸的快速检测。
其他相关产品线列表如下,欢迎订购!
| 通用编号 | 产品名称 | 品牌 | 规格 |
| C620035 | 重组 LbCas12a (Cpf1) | BBI | 100 pmol/1000 pmol |
| C620036 | 重组LwaCas13a (C2c2) | BBI | 100 pmol/1000 pmol |
| C620037 | 重组AapCas12b (C2c1) | BBI | 100 pmol/1000 pmol |
| C620038 | 重组Un1Cas12f1 (Cas14a1) | BBI | 100 pmol/1000 pmol |
| C620039 | 重组AsCas12a (Cpf1) | BBI | 100 pmol/1000 pmol |
| C620040 | 重组AacCas12b (C2c1) | BBI | 100 pmol/1000 pmol |
| C620041 | 重组FnCas12a(Cpf1) | BBI | 100 pmol/1000 pmol |
| C620042 | 重组Lb5Cas12a (Cpf1) | BBI | 100 pmol/1000 pmol |
参考文献
[1] Makarova Kira S, Wolf Yuri I, Iranzo Jaime, Shmakov Sergey A, Alkhnbashi Omer S, Brouns Stan J J, et al. Evolutionary classification of CRISPR-Cas systems: a burst of class 2 and derived variants. Nat Rev Microbiol. 2020 Feb;18(2):67-83. doi: 10.1038/s41579-019-0299-x. PMID: 31857715 PMCID: PMC8905525
[2] Yuye Wu, Dheerendranath Battalapalli, Mohammed J Hakeem, et al. Engineered CRISPR-Cas systems for the detection and control of antibiotic-resistant infections. J Nanobiotechnology. 2021 Dec 4;19(1):401. doi: 10.1186/s12951-021-01132-8. PMID: 34863214 PMCID: PMC8642896
[3] Feng Wei, Newbigging Ashley M, Tao Jeffrey, Cao Yiren, Peng Hanyong, Le Connie, Wu Jinjun, Pang Bo, et al. CRISPR technology incorporating amplification strategies: molecular assays for nucleic acids, proteins, and small molecules. Chem Sci. 2021 Mar 2;12(13):4683-4698. doi:10.1039/d0sc06973f. PMID: 34163728 PMCID: PMC8179559
.png)

