在基因表达的精密舞台上,转录与翻译如同 “生命剧本” 的两大核心篇章。长久以来,科学家对转录过程的探索早已细致入微,但翻译这个 “幕后关键环节” 却始终蒙着层神秘面纱 ——mRNA 的数量再多,也不等于蛋白质的合成效率;翻译的启停节奏、速率波动这些动态 “演技”,更是难觅踪迹。
直到 Ribo-seq(核糖体印记测序)技术诞生,才能以单碱基的精度,实时 “抓拍” 核糖体在 mRNA 上的每一步 “走位”,让翻译过程的复杂调控与精密协作首次暴露在视野中。这一技术不仅为分子生物学、医学和合成生物学带来了颠覆性的研究范式,更像一把金钥匙,为我们打开了理解基因表达的全新窗口。那么,这把 “金钥匙” 如何巧妙串联起多组学数据,成为解码生命奥秘的关键纽带?今天,小编就带大家解锁基因表达的 “隐藏视角”!
Ribo-seq+转录组学
通过对比 mRNA 丰度(mRNA-seq)与核糖体结合效率(Ribo-seq),可以明确 mRNA 转录水平与蛋白质合成效率的关联性,区分转录调控与翻译调控的贡献。鉴定出的翻译调控事件,又可以发现转录组未体现的翻译起始位点、上游开放阅读框(uORF)及翻译效率差异,挖掘隐藏的调控机制。从而帮助研究者整合转录层面( mRNA 丰度)和翻译层面(核糖体结合效率)数据,构建更完整的基因表达调控路径,并且还能筛选出功能性候选基因,从 “高转录 - 低翻译” 或 “低转录 - 高翻译” 的差异基因中,锁定受翻译调控的关键功能基因,用以验证转录本的翻译活性,区分有翻译潜力的功能性 mRNA 与无翻译活性的非编码转录本,排除假阳性蛋白编码基因。在时空或处理条件下,同步追踪 mRNA 转录变化与核糖体结合动态,揭示基因表达的动态表达调控机制。
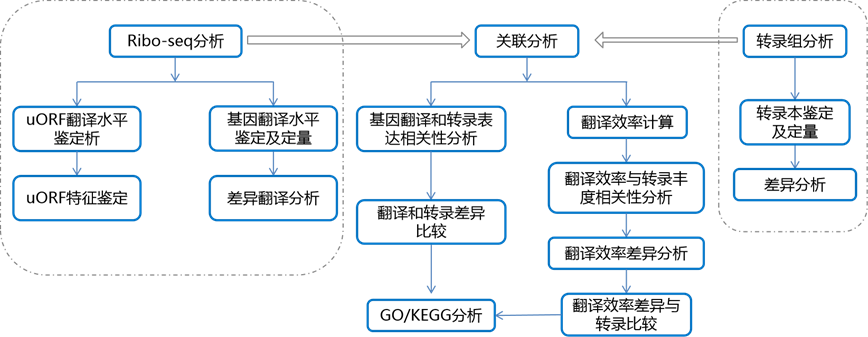

中文名称:人大脑发育过程中RNA翻译的动态变化【1】
发表时间:2022 年 9 月 28 日
发表期刊:Nature Neuroscience(IF=25)
研究背景:基因表达的精确调控对神经发育、可塑性和认知功能至关重要。此前许多研究聚焦于人类大脑发育中的基因组和转录组调控,但对翻译调控的复杂性了解较少,人类大脑中微蛋白的特性和作用几乎完全未被表征。
实验设计:该研究对 70 多个人类 prenatal 和成人皮层样本进行核糖体图谱(Ribo-seq)和 RNA 测序(RNA-seq),绘制翻译事件;同时对人类胚胎干细胞衍生的神经元培养物进行核糖体图谱分析,探究神经元活动响应的翻译变化;还结合大小选择蛋白质组学验证部分翻译产物,通过基因组系统发育分析 sORFs 的进化起源,利用物理化学分析预测微蛋白功能,借助 CRISPR-Cas9 扰动数据验证部分 sORFs 功能,通过分层连锁不平衡分数回归分析探究 sORF 变异与神经精神疾病的关联。
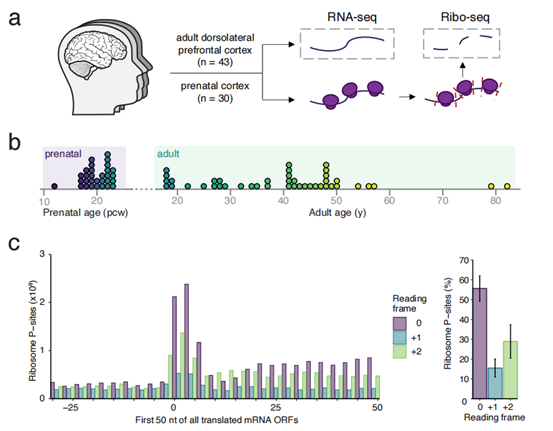
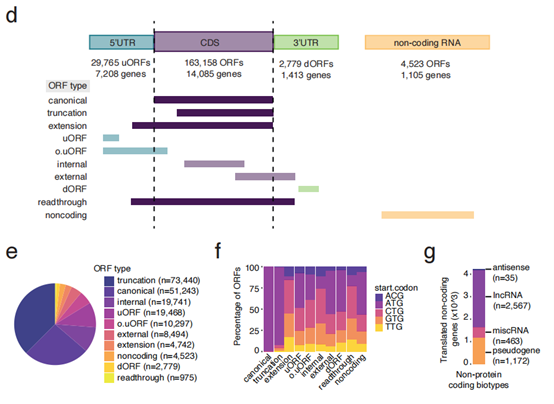
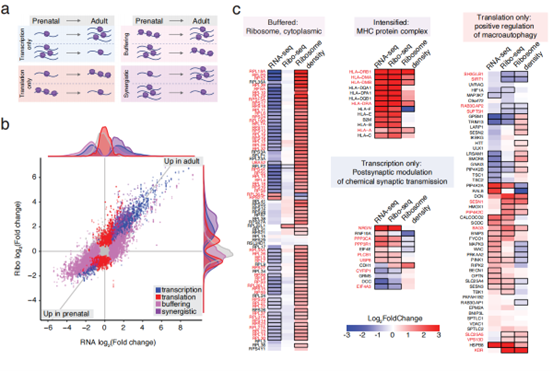
研究者通过分析大脑皮质样本的 Ribo-seq 数据,利用三碱基周期特征定位活跃翻译的核糖体,共识别出 172,187 个活跃翻译的开放阅读框(ORFs),映射至 13,305 个基因。研究重点聚焦于小开放阅读框(sORFs),从中鉴定出 8,278 个基因来源的 38,187 个活跃 sORFs,其中多个 sORFs 源自先前被注释为非编码的转录本。为验证这些 sORFs 的翻译活性,研究者将质谱数据与 Ribo-seq 结果匹配,在蛋白质水平上独立证实了其翻译产物的存在。同时,研究者探讨了上游开放阅读框(uORFs)对典型 ORFs 翻译的调控作用,发现部分 uORFs 与下游典型 ORFs 的翻译呈负相关。通过对 sORF 的序列分析,研究者预测了其编码微肽的理化特性,并推测这些微肽可能通过与 RNA 加工复合物相互作用,参与细胞核内 mRNA 剪接、翻译调控或 DNA 损伤反应。
该研究揭示了人类大脑翻译组的复杂性,指出非典型 ORFs 在大脑发育与疾病中的潜在作用,为后续探究神经系统翻译调控机制及新发现的人类大脑特异性微肽功能提供了重要资源与研究方向。
Ribo-seq+ncRNA组学
非编码 RNA(ncRNA)曾被视为“基因组暗物质”的翻译元件,但其在基因表达调控中的复杂作用正逐步被揭示。核糖体图谱技术(Ribo-seq),为解码 ncRNA 隐藏的翻译潜能提供了突破性工具 —— 通过捕捉核糖体结合的 RNA 片段,该技术能直接定位活跃翻译的区域,让那些被定义为 “非编码” 的转录本中潜藏的开放阅读框(ORFs)浮出水面。 这种联合分析不仅颠覆了对 ncRNA 功能的传统认知,更揭示出一个庞大的 “暗翻译组”重新定义基因组的蛋白质编码边界,为探索生命活动的分子机制开辟了全新维度。


中文名称:长链非编码 RNA 的保守区域包含丰富的翻译和蛋白质 - RNA 相互作用特征【2】
发表时间:2019 年 7 月 5 日
发表期刊:《NAR Genomics and Bioinformatics》(IF=11.147)
研究背景:哺乳动物转录组中包含数千个不属于注释的蛋白质编码基因的转录本,即长链非编码 RNA(lncRNAs)。目前仅有少数 lncRNAs 的调控功能被充分阐明,绝大多数 lncRNAs 的生物学意义尚不明确。在小鼠和人类之间保守的 lncRNAs 可能富含功能序列。核糖体图谱技术(Ribo-Seq)的出现为研究 lncRNAs 的翻译情况提供了有力工具,该技术能识别核糖体保护的 RNA 片段,其读取覆盖度可提供蛋白质翻译的明确信号。同时,lncRNAs 与蛋白质形成核糖核蛋白复合物(RNPs)的相互作用也是其功能研究的重要方面。在此背景下,研究旨在探究 lncRNAs 中不同类型的核糖体图谱特征及其与序列保守性的关系,以深入了解 lncRNAs 的功能景观。
实验设计:从 Ensembl v.89 获取小鼠序列及调控区域,经筛选和重复序列屏蔽后,结合 BLASTN 识别小鼠与人类转录本的保守区域;采用小鼠海马体的 RNA-Seq 和 Ribo-Seq 数据,结合 RibORF 预测翻译 ORFs,借助质谱数据及 dN/dS、PhyloP 分析验证其功能性;运用 Rfoot 和 FLOSS 方法预测 RNP 位置,并结合 CLIP-seq 数据集验证,进而分析不同区域的 RNP 特征,同时还对基因进行分类以探究不同类型 lncRNAs 的特征。
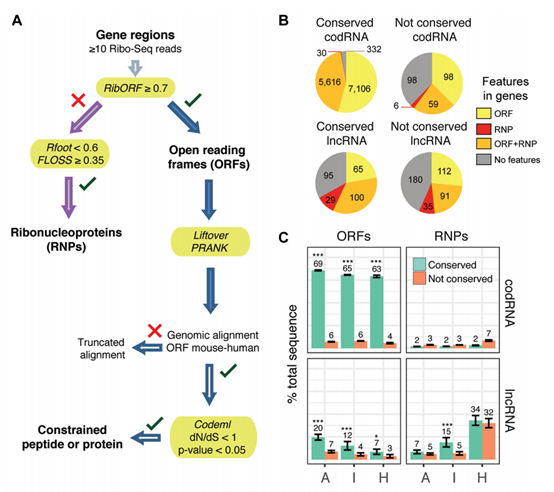
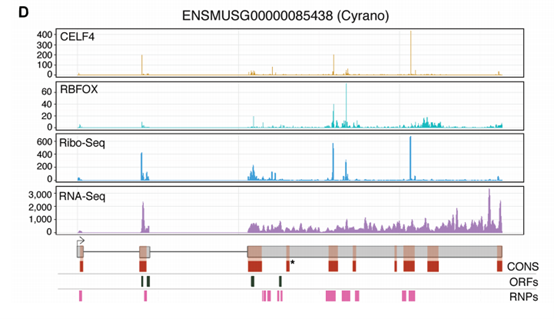
该研究发现,小鼠和人类之间保守的长链非编码 RNA(lncRNAs)区域富含翻译的开放阅读框(ORF)和蛋白质 - RNA 相互作用(RNPs),其中保守区域的翻译 ORF 数量约是非保守区域的三倍,且在不同 lncRNA 亚型中均存在此趋势;保守 lncRNAs 受到纯化选择作用,进化速率低于中性进化预期;在 lncRNA 保守区域的翻译 ORF 中鉴定出 9 个可能编码受选择约束的功能性蛋白质的案例;通过 Ribo-Seq 数据等预测出 lncRNAs 中的 RNPs,保守区域的 RNPs 数量多于非保守区域,在已知和未知功能的 lncRNAs 中均有发现,为探索 lncRNA 功能提供了新候选区域。
Ribo-seq+RNA甲基化
国自然热点联动,揭示RNA修饰对翻译的动态调控
Ribo-seq 与 RNA 甲基化的交叉研究已成为生命科学领域的新兴前沿,在 RNA 甲基化修饰中,m6A 修饰因其广泛的生物学功能而备受关注 —— 它可通过直接作用(如改变 mRNA 二级结构以影响核糖体结合)或间接调控(如招募甲基化结合蛋白介导翻译起始调控),动态调节核糖体与 mRNA 的结合效率及翻译进程,最终导致 Ribo-seq 技术所捕获的核糖体保护片段(RPFs)在分布模式与丰度水平上产生特征性差异。作为解析翻译动态的核心技术,Ribo-seq 为系统探究 RNA 甲基化的翻译调控功能提供了关键实验支撑:通过对甲基化与非甲基化 RNA 的 Ribo-seq 数据进行对比分析,能够精准筛选出受甲基化修饰调控的靶标 RNA,并量化其翻译效率的改变,进而阐明 RNA 甲基化在特定生理活动或病理过程中的分子作用机制。


中文名称:多组学数据研究人类巨噬细胞分化和极化中的RNA修饰【3】
发表时间:2024年2月
发表期刊:Scientific Data(IF=9.8)
研究背景:RNA 修饰作为基因表达调控的核心机制,其中 N⁶- 甲基腺苷(m⁶A)和 5 - 羟甲基胞嘧啶(5hmC)备受关注。m⁶A 通过 “写入”“擦除”“读取” 蛋白动态调控 RNA 代谢,参与细胞发育、免疫应答等生理过程,而 5hmC 的功能研究相对有限,仅少数研究涉及转录组层面的作用。巨噬细胞作为免疫防御的关键细胞,具有高度可塑性,可分化为静息态或极化为促炎(M1 样)、抗炎(M2 样)表型,但其分化与极化过程中 RNA 修饰(尤其是 m⁶A 和 5hmC)对基因表达的调控机制尚未明确。因此,构建涵盖巨噬细胞分化与极化过程的多组学数据集,对解析 RNA 修饰的作用具有重要意义。
实验设计:研究者以 THP-1 单核细胞为模型,通过体外诱导构建四种细胞状态:单核细胞、静息态巨噬细胞、促炎样巨噬细胞和抗炎样巨噬细胞。对四种细胞进行多组学检测,转录组(mRNA-Seq)、表观转录组(m⁶A-IP-Seq 和 5hmC-IP-Seq)、翻译组(Polyribo-Seq)和蛋白质组(LC-MS/MS)。将同一份RNA 样本同步用于 mRNA-Seq、m⁶A-IP-Seq 和 5hmC-IP-Seq检测,以确保数据整合性并设置生物学重复,通过质量控制验证数据可靠性,再结合已知标志物和修饰特征(如 m⁶A 的 DRACH 基序、5hmC 的 UC 富集 motif)验证生物学一致性。
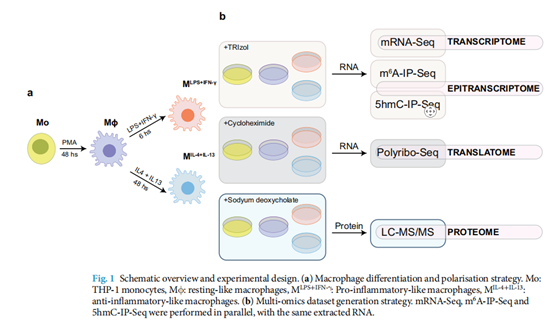
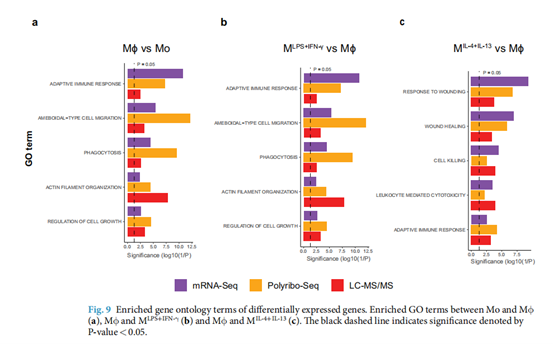
研究构建了包含 mRNA-Seq、m⁶A-IP-Seq、5hmC-IP-Seq、Polyribo-Seq 和 LC-MS/MS 的多组学数据集,覆盖单核细胞及静息态、促炎样、抗炎样巨噬细胞这四种细胞状态,全面展现巨噬细胞分化与极化过程中的基因表达调控情况。通过对这些数据的分析,揭示了 m⁶A 和 5hmC 这两种 RNA 修饰在巨噬细胞生物学中的作用机制。同时,研究还验证了不同组学数据样本间的高质量与相关性,以及模型巨噬细胞在转录组、表观转录组、翻译组和蛋白质组水平的生物学一致性,为深入探究 RNA 修饰在巨噬细胞发育及功能方面的影响提供了宝贵资源 。
综上所述,Ribo-seq 在医学领域早已搭建起从翻译效率动态追踪到跨组学机制深挖的成熟研究框架。熟悉该技术的研究者可借鉴这些范式与策略,助力自身研究开展哦~
如果老师对转录组,蛋白组,翻译组感兴趣,尽快联系我们吧,做多组学项目的话,还可以赠送联合分析吆~~
技术支持
电话:021-57072059/2097/2177/2179
邮箱:RNAseq@sangon.com/RNAseq2@sangon.com/RNAseq3@sangon.com
【1】Duffy E E, Finander B, Choi G H, et al. Developmental dynamics of RNA translation in the human brain[J]. Nature Neuroscience, 2022, 25(9):1333 - 1345. DOI:10.1038/s41593-022-01174-4
【2】Ruiz - Orera J, Alba MM. Conserved regions in long non - coding RNAs contain abundant translation and protein–RNA interaction signatures[J]. Nucleic Acids Research, 2019, 47(14):7321 - 7335. DOI:10.1093/nar/gkz361
【3】Pinello N, Song R, Lee Q, et al. A multiomics dataset for the study of RNA modifications in human macrophage differentiation and polarisation[J]. Scientific Data, 2024, 11(1):252. DOI:10.1038/s41597-024-03076-8.

