生工技术 | molbiotools功能大赏
molbiotools(www.molbiotools.com)是一个关于DNA序列处理、数据处理以及实验相关计算的综合性的辅助网站,由几个独立工具组成。
打开该网站,一阵简约风扑面而来,网站简单到连个自我介绍都没有,唯独能够看出来的只有最下方的© 2020 Vladimír Čermák. (暗示着网站的作者?)

从上面的概览图也可以看到,网站由三大模块组成,每个模块集成了6个功能,它们分别是:

但不要被网站的简约外表欺骗,打开其中的工具,会发现该网站简约而不简单。其中有几个功能还是非常实用的,在这里推荐给大家。
WebDSV——DNA序列编辑器和查看器
日常实验中,我们经常会在一些DNA序列上进行标记,比如记录目的基因的位置,引物的序列及位置等,比如:

还在word中编辑自己的DNA序列吗?Out了,买不起SnapGene的我们有了新的选择,打开www.molbiotools.com/WebDSV/,映入眼前的是一个专业的序列编辑和标注窗口,所有的序列查看和备注都可以在这一个窗口中完成:
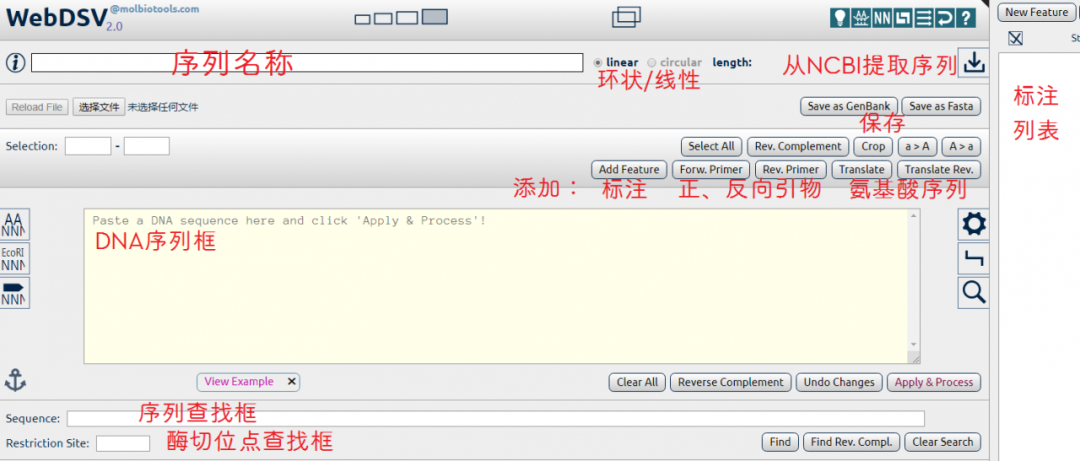
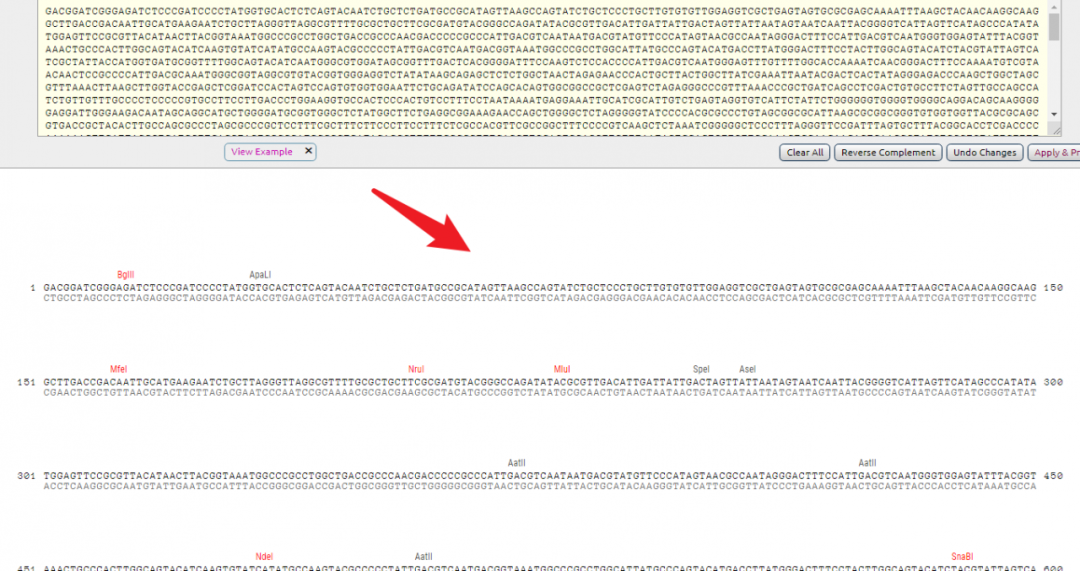
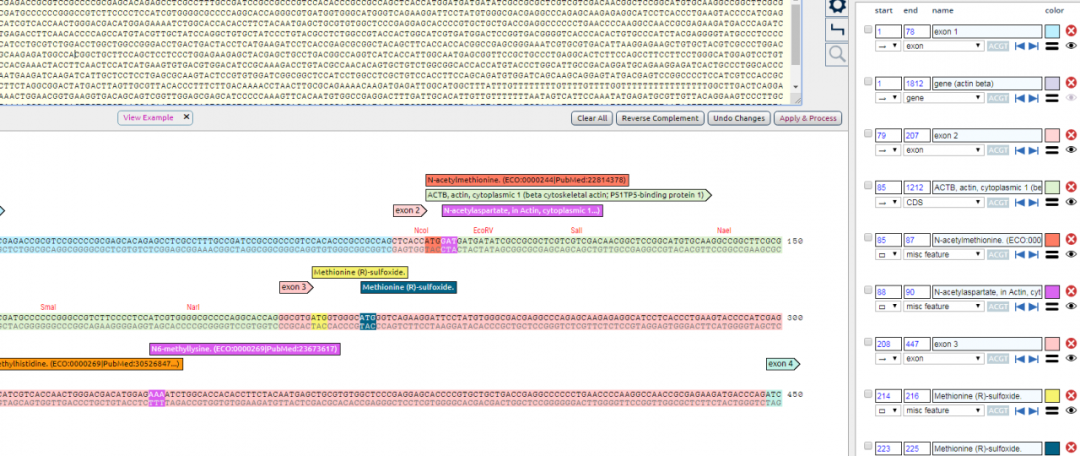
1. 输入名称和序列,点击Apply & process 确认按钮,会看到,下方会出现该序列的可视化图形,并且一些常用的酶切位点已经标注在了序列上:
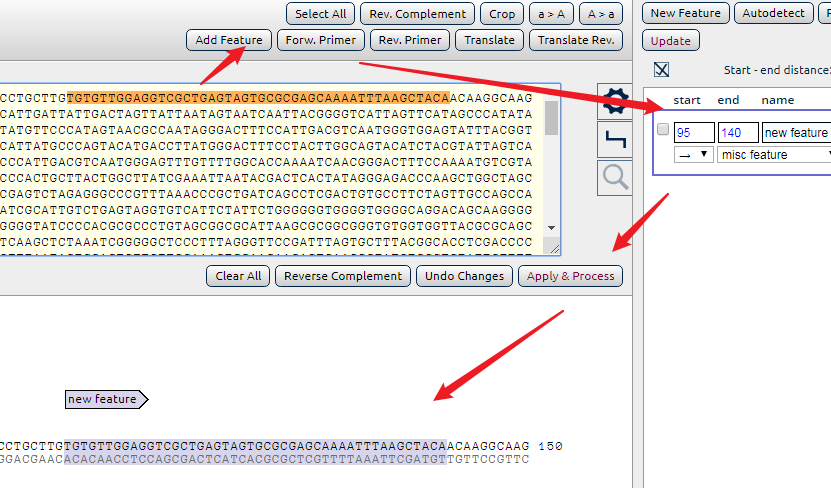
2. 在上方序列框中选中目的序列,点击add feature(添加标记),会在右侧标记列表中新增这段序列的标记选项,设置好之后,点击确认按钮,会发现在下方的可视化界面中相应的区段已经添加该段标记。
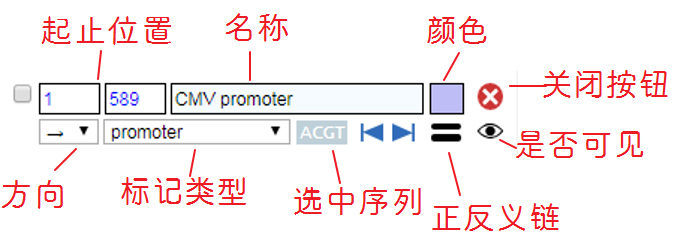
Feature标记的设置选项如下:
3. 同样的处理方法,还可以将自己设计的引物,添加到序列中:
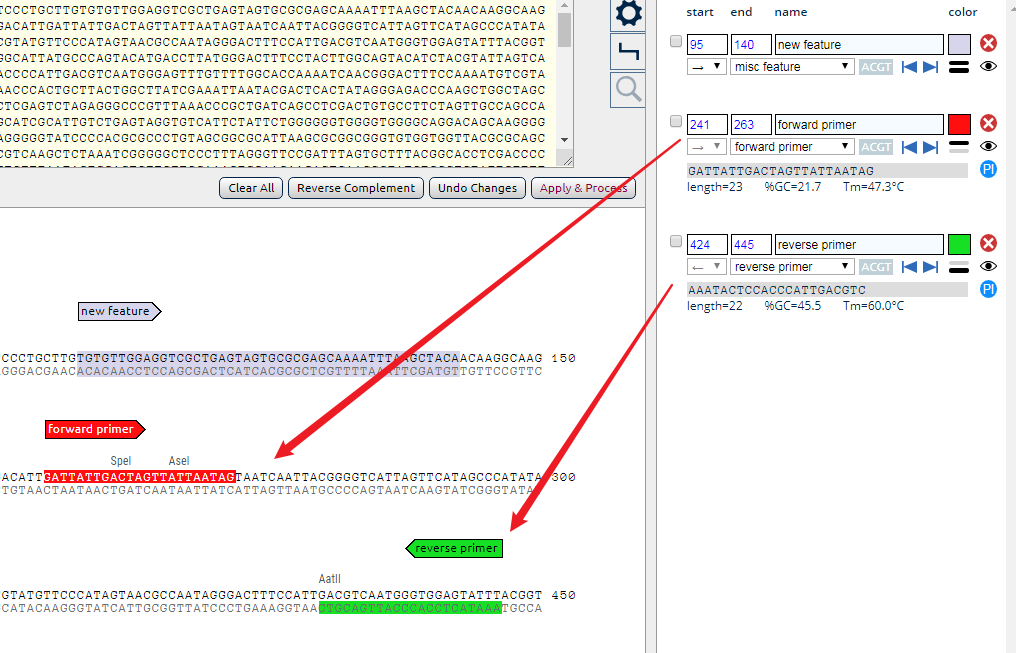
4. 如果输入的序列中含有常见的序列,比如一些复制子、启动子、抗生素耐药基因等,点击右上角AutoDetect,系统就会自动标记,比如,我将pegfp-N1的质粒序列粘贴进去,其自动识别出了其大多数基因元件。
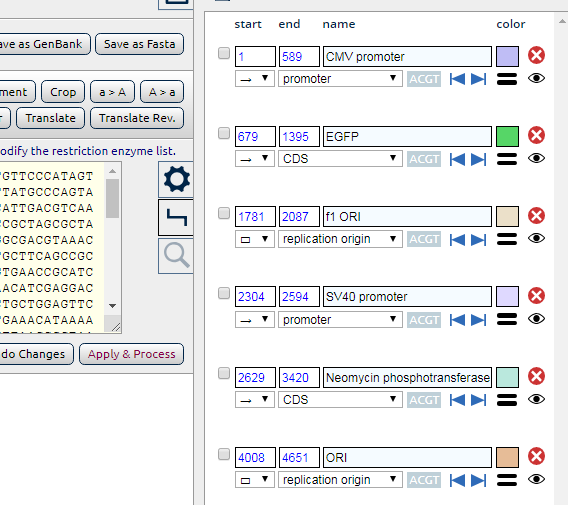
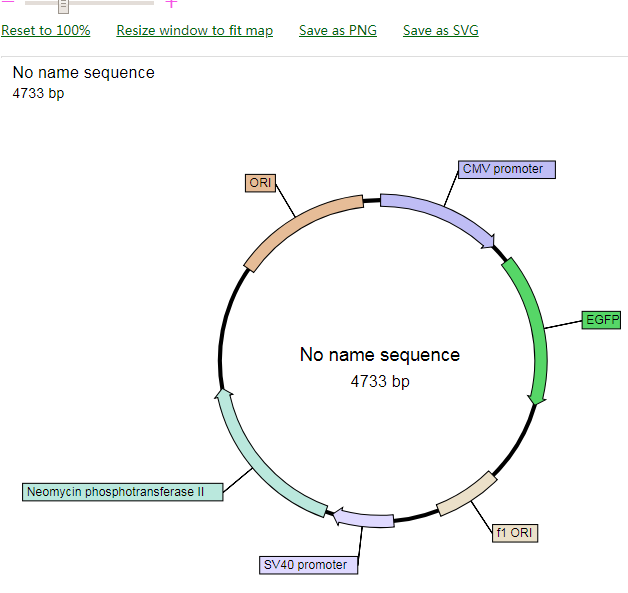
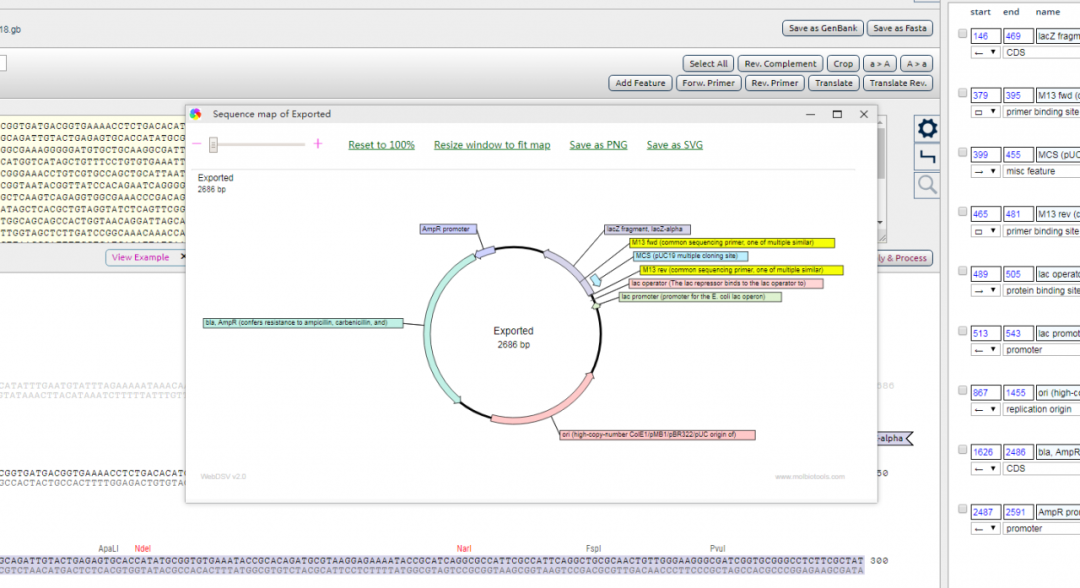
5. 自动生成图形,点击右上角的Map,网页会新弹出一个窗口,显示现在的基因结构及我们添加的标记,酶切位点等
6. 一切都设置好了之后,可以点击Save as GenBank
从而生成一个gb文件,可以用记事本打开该文件查看序列或标记,也可以用WebDSV重新打开该gb文件来继续可视化或编辑。

7. 如果您要标记某个基因,那么更简单了,直接点击这个按钮,
将基因的accession号输入进去,比如NM_001101,点击Download,系统会自动从NCBI将相关accession编号的序列和相关标注信息导入进来。非常的方便。
实际上,任何形式的GenBank文件,都可以直接用这个工具导入,并立即进行可视化:
PCR Primer Inspector——PCR引物检查器
PCR引物检查器(PCR Primer Inspector)是用于计算PCR引物的预测熔解温度,并分析其在PCR反应条件下形成同源或异源二聚体的倾向。在做PCR尤其是多重PCR的时候,引物之间的相互结合扩增,可能会对最终结果产生不良的影响,因此在进行实验之前可以用这个工具预测一下引物之间相互结合的可能性。
检测方法很简单,将PCR体系中的引物序列按照分行的形式添加到序列框中即可,比如下面几条引物

点击Apply & Analyze,出现如下几个结果,根据结合位置的不同,分为不同的处理情况:

引物自身或者引物之间的3端互补配对,这是最不好的情况,因为这种情况会导致结合之后的引物3端产生延伸,从而导致二聚体产物的出现,遇到这种情况必须对引物进行优化处理。
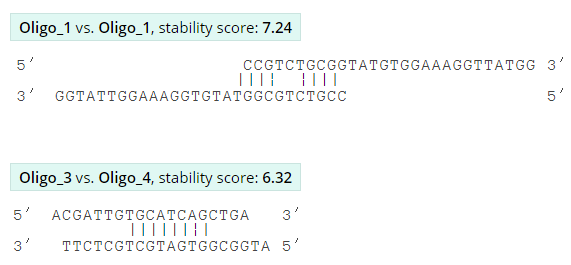
上图这两种情况分别是引物的5端配对或者中间配对,它们有个共性,就是3端都是翘起的,没有参与互补配对,这两种情况通常情况下不会对结果产生影响,因为即使在退火过程中引物结合到了一起,也不会实现扩增。
Restriction Comparator
限制性酶切位点比较器
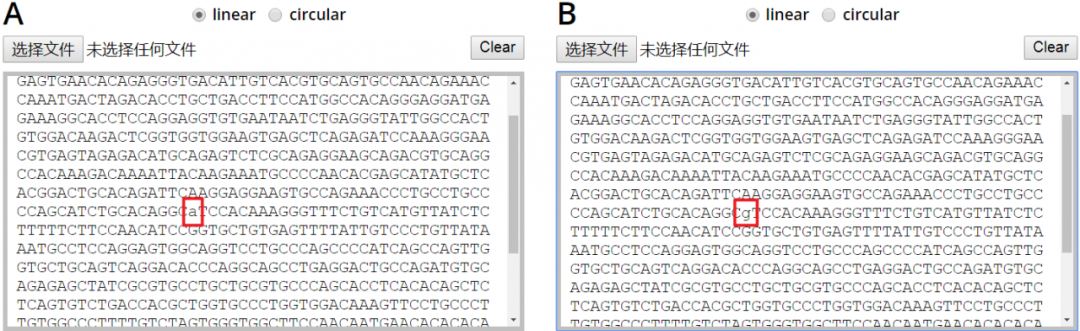
Restriction Comparator用于两个DNA序列A、B的限制性酶切位点比较分析。它可以检测限制性内切酶位点,这些限制性内切酶位点存在于一个序列中但不存在于另一个序列中,或具有不同频率。此外,它可以提供DNA使用内切酶进行酶切之后的电泳图模拟,并排显示两个序列的限制性片段模式。
该功能可以用于SNP位点的PCR-PFLP的实验方案设计,比如检测rs9462082(NC_000006.11:g.35386041A>G)突变,将SNP位点及其侧翼序列分别粘贴到左右两个序列框中。注意,两个序列分别将SNP位点设置为不同的基因型,左侧可以设为A,右侧设为G。
右侧的设置栏中,我们勾选上所有的限制性酶种类,并将酶切之后的片段大小差异最小值设为0,
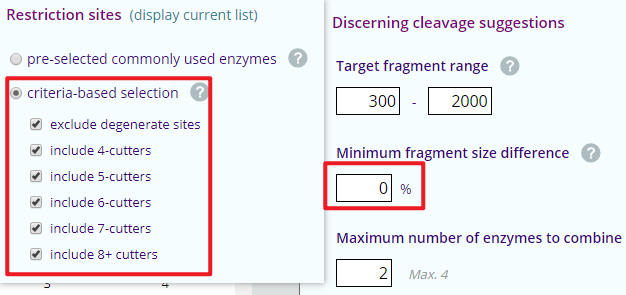
点击Apply & Analyze,会得到以下结果:
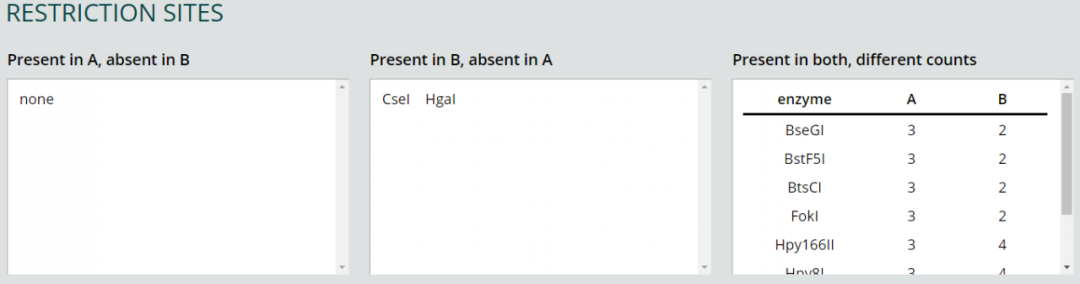
三个结果中分别显示了只存在于序列A,只存在与序列B与同时在两个序列中都存在的酶切位点。由于序列A和B唯一的区别就在于SNP位点,因此我们就可以利用仅存在于一条序列中的酶切位点CseI或HgaI对该序列进行RFLP分析。
接下来,通过调整序列A和B的长短,我们在下方酶切列表中选择选中的酶CseI,该工具会自动显示出模拟的酶切电泳图,非常直观。
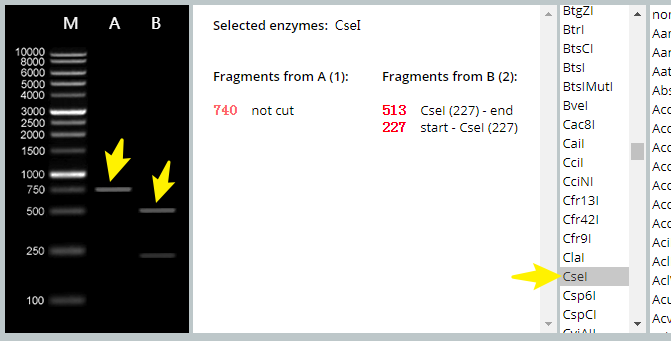
该工具除了帮助我们设计RFLP实验方案,还有其他用途,感兴趣的可以尝试一下。
DNA Calculator——DNA计算器
使用该工具,可以对引物、质粒等单链或者双链的DNA的一些性质进行计算。
比如,输入序列TATCTAGCTAGCTCTAGCTAGC,然后设置下方参数:

计算出结果如下,与生工生物的Oligo计算器相比,结果是几乎一致的。
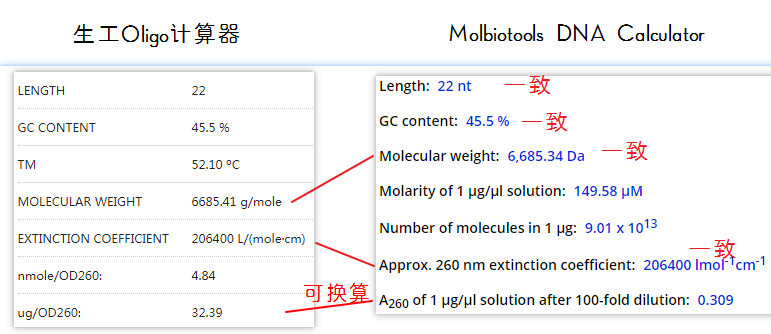
除了对Oligo进行计算,还可以对质粒这种长的双链DNA进行计算,比如在做定量PCR时,需使用阳性质粒配制不同浓度梯度的溶液。使用该工具,可以轻松计算出1ug质粒对应的拷贝数:
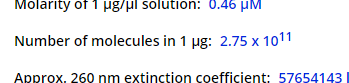
抑或是测浓度时,根据A260的吸光值计算出质量浓度:

抑或是该加多少水来配制成想要的浓度:

Chemical Calculator——化学计算器
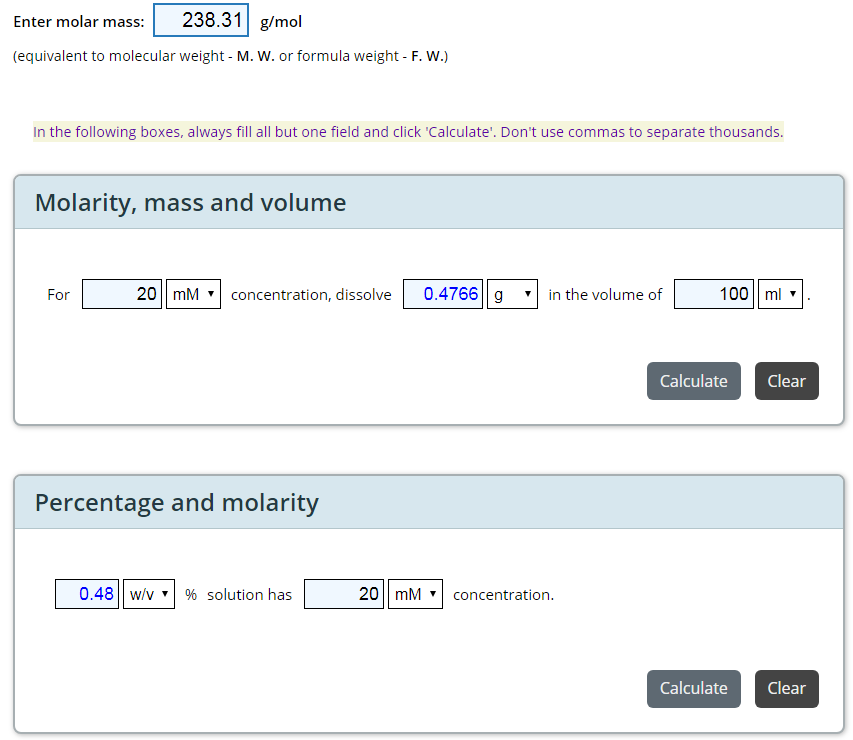
有了它,配制化学品溶液将变得很简单,再也不用计算器算来算去了,比如,要配制100 ml浓度为20 mM的IPTG溶液,该称取多少IPTG粉末呢?
将IPTG的分子量以及浓度或体积输入进去,自动会计算出0.4766 g。
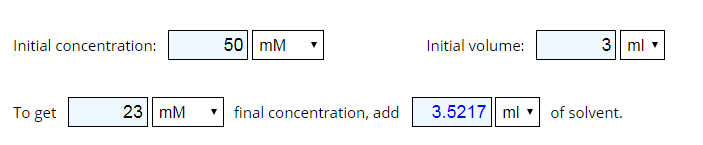
此外,还可以计算母液如何稀释成工作液,或者稀释成其他想要的浓度等。
以上就是小编经常使用的功能推荐,此外,Molbiotools中还有其他一些功能,与数据处理、分析相关,各位感兴趣的小伙伴可以前往探索。
网址:www.molbiotools.com

