生工技术 | 碱基淬灭探针
只有荧光基团没有淬灭基团的探针也会有荧光值的变化?
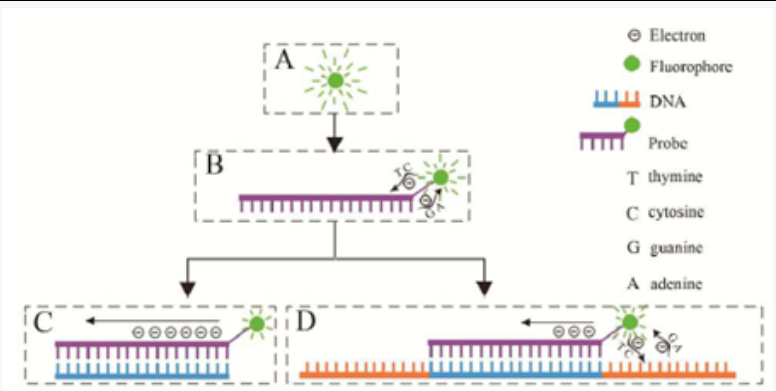
我们知道,在经典的Taqman探针法中,Taqman探针的两端分别标记荧光基团和淬灭基团,由于Taq酶均有5’-3’核酸外切酶活性,在引物的延伸过程中首先发生链置换,后续发生探针剪切。剪切使得荧光和淬灭基团相互分离,释放荧光信号。在每一轮的延伸反应下,Taqman探针都会被剪切,导致荧光信号不断的积累[1]。

如果在探针上只标记一个荧光基团,那么参考Taqman探针的原理,不管探针有没有被Taq酶切断,由于没有淬灭基团的关系,荧光信号的强度理论上是不会发生改变的。那么它是怎么实现荧光信号的检测呢?
这还得从Taqman探针设计中的禁忌谈起,如果Taqman探针中荧光基团相邻的碱基为G,将会使一部分的荧光信号被淬灭,最高可能达到40%左右。
因此Taqman探针中荧光基团相邻的碱基会尽量避免设计为G。
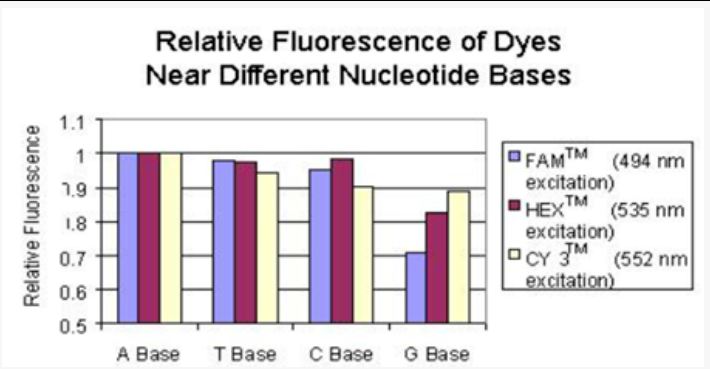
图片来自于IDT DNA
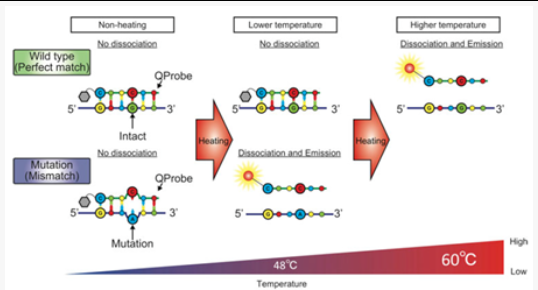
正是利用了G碱基对荧光基团的淬灭效果,只标记一个荧光基团的探针也可以完成SNP的检测,它的正式名称为碱基淬灭探针[2]。碱基淬灭探针的检测不需要借助Taq酶的5’-3’外切酶活性,而是在所有延伸都完成后,探针和PCR产物进行溶解曲线分析。溶解曲线分析的初始温度一般为40 ℃,终点温度可以在80 ℃左右。
碱基淬灭探针的5’末端第一个碱基为C碱基,与荧光基团相邻。当探针与PCR产物结合时,由于G碱基对荧光基团的淬灭效果,荧光信号下降。随着溶解曲线分析中温度的升高,突变型的PCR产物因为和探针有一个错配,探针在较低的温度就会与PCR产物发生分离;野生型的PCR产物与探针是完全匹配的,在较高的温度才会与PCR产物发生分离[3]。
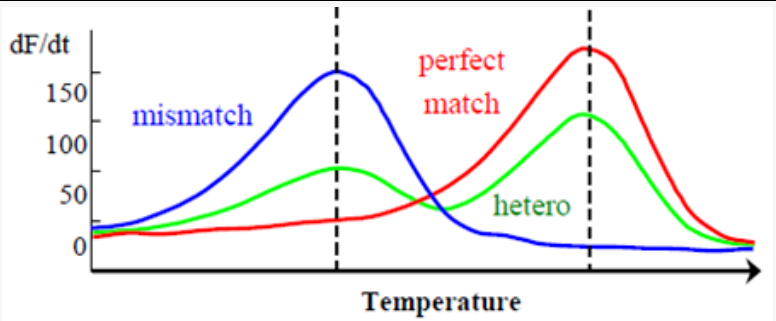
PCR产物与探针发生解离时,荧光信号上升,以荧光信号的导数(荧光信号的变化速率)为纵坐标,以温度为横坐标,即可得到下图的溶解曲线结果,蓝色曲线为突变型,红色曲线为野生型,绿色曲线为杂合子。通过对溶解曲线中山峰的位置,实际上就是探针与PCR产物结合时的Tm值,即可对SNP情况进行判断。
后续的研究表明,不只是G碱基,ATC三种碱基也会对荧光产生一定的淬灭作用,使用这三种碱基也可以完成对SNP的检测。且碱基对荧光基团的淬灭不仅限于FAM,HEX, CY5, CY3, TET, JOE, Texas Red和ROX这些荧光基团都能被碱基淬灭[4]。只是效果不如G碱基这么明显[5]。看到这些文章的时候,小编也十分惊讶,碱基可以淬灭荧光?

那么,碱基是怎么完成荧光淬灭的过程呢?Taqman探针中淬灭基团是通过FRET吸收了荧光基团发出的荧光。碱基对荧光的淬灭则是一种被称为光诱导电子转移(photoinduced electron transfer, PET)产生的[6]。这个词可能你完全没有接触过,没关系,下面的内容小编来进行解释。
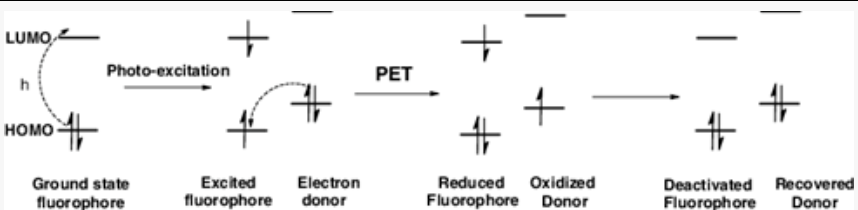
在受到光激发时,荧光基团中处于基态的部分电子吸收了光的能量跃迁到激发态,在从激发态(严格来说是第一激发态)回到基态时,能量会以光的形式释放出来,这就是荧光。在单链DNA中,荧光团的电子被转移到嘧啶碱基的轨道上,或者荧光团的电子轨道被来自嘌呤碱基的电子占据,因此荧光团的电子无法还原到基态,导致荧光猝灭。除此之外,荧光基团中的电子会沿着探针DNA链和模板链的碱基进行电子转移,这样荧光基团可用的电子数减少,也会淬灭部分荧光[7]。
看到这里,我相信会有一票人跳出来说,小编你是不是有点扯,单标记荧光的DNA链也能检测到荧光啊,没有你提到的淬灭吧。这里小编要说明的是,这里的荧光淬灭,并不是让荧光完全消失看不到,以BHQ类淬灭基团为例,实际上它淬灭效率一般在80-98%之间[8],并非是100%的淬灭荧光。之前我们提到的G碱基对荧光的淬灭效率,最高也就是40%。至于普通的碱基ATC,对于荧光的淬灭效率就更低了。因此这种荧光的变化需要比较精密的仪器才能较好的测量,从小编看到的文献来说,碱基淬灭探针很多用到的是Roche LightCycler 480荧光定量PCR仪。
碱基淬灭探针可以使用带有弱的5’-3’外切酶活性的Taq酶用于扩增,上下游引物的终浓度一般为0.5 μM,探针的终浓度为0.1 μM。扩增程序为95 ℃预变性2 min,95 ℃变性2 s,58 ℃退火 10 s,60 ℃延伸 1min,变性,退火,延伸进行40个循环。溶解曲线分析步骤为:95 ℃ 30 s,25 ℃ 4 min,升温到 80 ℃,升温速率0.1 ℃/s。
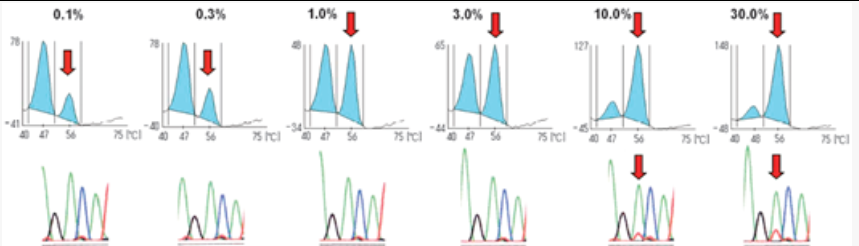
下图为使用碱基淬灭探针检测c-kit外显子17上的D816V突变的结果。使用突变片段和正常受试者DNA混合物进行分析,分别对应于0.1%,0.3%,1%、3%,10%和30%稀释度的样品进行灵敏度分析。相比基础的测序办法,碱基淬灭探针具有很高的灵敏度,即使0.1%突变模板也能被检测出来[9]。
1.Anton A. Komar. Single Nucleotide Polymorphisms. Methods and Protocols.
2.Crockett AO, Wittwer CT. Fluorescein-labeled oligonucleotides for real-time pcr: using the inherent quenching of deoxyguanosine nucleotides. Anal Biochem. 2001 Mar 1;290(1):89-97.
3.Kurimoto M, Suzuki H, Aoki K, Ohka F, Kondo G, Motomura K, Iijima K, Yamamichi A, Ranjit M, Wakabayashi T, Kimura S, Natsume A. Rapid sensitive analysis of IDH1 mutation in lower-grade gliomas by automated genetic typing involving a quenching probe. Cancer Invest. 2016;34(1):12-5.
4.Mao H, Luo G, Zhang J, Xu N. Detection of simultaneous multi-mutations using base-quenched probe. Anal Biochem. 2018 Feb 15;543:79-81.
5.Luo G, Zheng L, Zhang X, Zhang J, Nilsson-Ehle P, Xu N. Genotyping of single nucleotide polymorphisms using base-quenched probe: a method does not invariably depend on the deoxyguanosine nucleotide. Anal Biochem. 2009 Mar 15;386(2):161-6.
6.Aigner D, Borisov SM, Klimant I. New fluorescent perylene bisimide indicators--a platform for broadband pH optodes. Anal Bioanal Chem. 2011 Jun;400(8):2475-85.
7.Mao H, Luo G , Zhan Y , Zhang J , Yao S , Yu Y . The mechanism and regularity of quenching the effect of bases on fluorophores: the base-quenched probe method. Analyst. 2018 Jul 9;143(14):3292-3301.
8.Marras SA, Kramer FR, Tyagi S. Efficiencies of fluorescence resonance energy transfer and contact-mediated quenching in oligonucleotide probes. Nucleic Acids Res. 2002 Nov 1;30(21):e122.
9.Wakita S, Yamaguchi H, Miyake K, Mitamura Y, Kosaka F, Dan K, Inokuchi K. Importance of c-kit mutation detection method sensitivity in prognostic analyses of t(8;21)(q22;q22) acute myeloid leukemia. Leukemia. 2011 Sep;25(9):1423-32.

