CUT&Tag(Cleavage Under Targets and Tagmentation, CUT&Tag)技术,是用于研究蛋白质与DNA之间相互作用,并为感兴趣的蛋白质确定DNA结合位点的一种分子生物学方法。由Henikoff博士于2019年发表在Nature Communication上,文章详细介绍了该技术的原理和方法。该技术与传统CHIP-Seq技术相比,具有实验周期短、步骤简单(无需超声破碎和免疫共沉淀)、细胞起始量低(10万以下,乃至单细胞)、重复性好、信噪比高等优势,被称为“新一代CHIP-Seq技术”,让使用的样本细胞量从104级别降到60个细胞甚至单细胞。为科研工作者们提供了一个新的选择。
CUT&Tag技术的核心是Tn5转座酶与protein A/G结合形成的PAG-Tn5重组蛋白,同时该重组蛋白连接有建库接头引物,方便后续将切断的DNA片段直接建库。该重组蛋白依赖protein A/G的抗体结合功能域,通过抗原抗体识别的原理与染色质上目的蛋白结合,然后激活Tn5的转座酶活性,将目的蛋白附近的DNA片段打断,同时将携带的Adaptor序列相当于转座子的结构“跳跃”到被打断的DNA片段上,后续DNA片段因为携带建库接头引物,则可以直接进行建库测序操作,从而大大缩短实验周期。
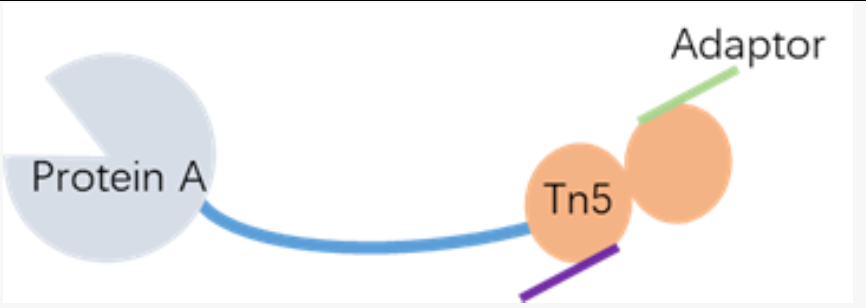
图1. PAG-Tn5重组蛋白的结构示意图
与CHIP-Seq的方法不一样的是CUT&Tag实验中的新鲜活细胞样本是固定于固相磁珠载体上进行反应的。不同于传统的离心收集,CUT&Tag利用刀豆蛋白能够与细胞膜上糖蛋白结合的原理,通过连有刀豆蛋白A的磁珠(concanavalin A beads)进行细胞的收集。利用磁珠结合细胞后,还需要使用非离子去垢剂洋地黄皂苷(digitonin)对细胞膜进行通透(原理是与胆固醇分子结合),为抗体等的进入提供可能性。
至此完成细胞样本的处理,后续的实验步骤都将发生于小小的磁珠上。
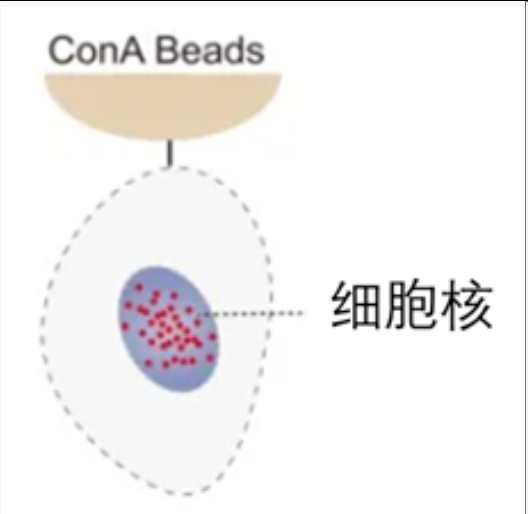
图2. 磁珠吸附细胞示意图
在处理好的细胞上,首先进行靶蛋白特异性抗体(一抗)孵育,使抗体进入细胞与靶蛋白结合。为了放大信号,同理接着进行二抗孵育。然后加入pAG-Tn5转座体进行孵育,使得转座体进入细胞并与二抗结合,这样就把转座体间接的固定在靶蛋白上。
另外需要注意的是pAG-Tn5转座体的激活依赖Mg2+,此时我们在反应体系中加入适量Mg2+激活Tn5酶的切割活性,在打断靶蛋白结合的DNA区域的同时将携带的建库接头转移到DNA片段上,之后提取DNA,进行PCR扩增构建文库,就可以进行测序啦~
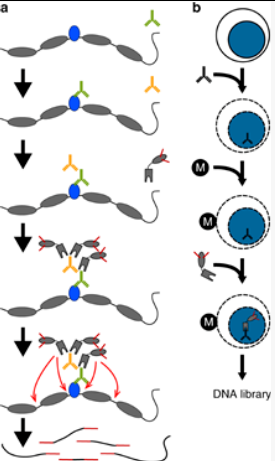
图3. 实验流程示意图[1]
a.CUT&Tag的步骤。
b. CUT&Tag在坚固的磁珠(M)上进行。
小伙伴们如果对Tn5转座酶到底是如何进行DNA片段切割以及测序接头转移的问题感兴趣,可以详细了解ATAC-seq技术原理哦~
表1. CUT&Tag与ChIP-Seq技术的对比
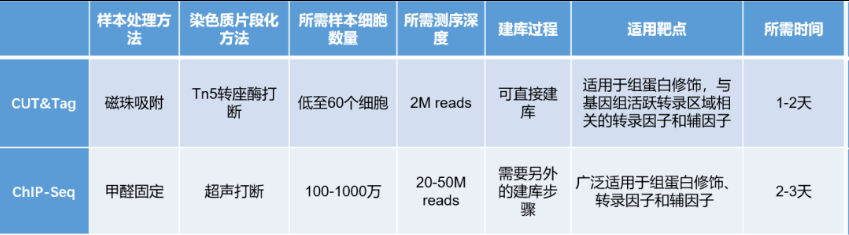
从上表中我们不难看出,CUT&Tag技术将带来更快速和准确的实验分析结果。
实验基于抗原抗体结合反应进行构建,不仅提高了特异性,采集到的数据背景噪音也更低,信噪比高,不需要input数据进行背景去除。

CUT&Tag的重复性更高,peak-calling的效率更高。
所需样本数量更少,科研人员使用较少的样本即可进行操作分析,并且处理方法简单,实验时间大大缩短。
CUT&Tag技术自诞生以来,就受到了广大科研工作者的喜爱,已有多篇文献证明其在不同的靶蛋白中均具有广泛的适用性。已有文章证实,CUT&Tag对RAR、CCKBR、TWIST2(转录因子)、JUN、JUNB、PIM1、Runx1等DNA直接或者间接结合蛋白都具有很好的适用性[2][3]。
针对不同的实验室模式生物CUT&Tag技术也都有很好的适用性,包括人、动物、植物、微生物样本等[4] [5] [6]。还有科研人员尝试了CUT&Tag在单细胞水平上的测序分析,实验结果证明CUT&Tag技术在单细胞测序方面也有很好的应用,而这也是ChIP-Seq技术所不能实现的。
实验室做不了?不要着急,小编带着生工生物CUT&Tag技术服务来咯,
生工生物CUT&Tag技术服务上线,您只需要提供符合实验的样本、检测目的蛋白的抗体,即可安心等待检测报告,还有专业的生物信息学数据分析结果。生工还提供用于实验的抗体,有需要的小伙伴也可以直接选择生工生物的抗体产品。
想了解更多详细信息,欢迎您随时和我们的技术支持联系,进行技术咨询
技术支持电话:021-57072054
邮箱:service8@sangon.com
点击链接了解更多详细信息吧~
https://www.sangon.com/services_Cut_Tag.html
参考文献
[1] Kaya-Okur, H.S., Wu, S.J., Codomo, C.A. et al. CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nat Commun 10, 1930 (2019).
[2] Bartosovic M, Kabbe M, Castelo-Branco G. Single-cell CUT&Tag profiles histone modifications and transcription factors in complex tissues. Nat Biotechnol. 2021 Jul;39(7):825-835. doi: 10.1038/s41587-021-00869-9. Epub 2021 Apr 12. PMID: 33846645; PMCID: PMC7611252.
[3] Yashar WM, Kong G, VanCampen J, Curtiss BM, Coleman DJ, Carbone L, Yardimci GG, Maxson JE, Braun TP. GoPeaks: histone modification peak calling for CUT&Tag. Genome Biol. 2022 Jul 4;23(1):144. doi: 10.1186/s13059-022-02707-w. PMID: 35788238; PMCID: PMC9252088.
[4] Gopalan S, Fazzio TG. Multi-CUT&Tag to simultaneously profile multiple chromatin factors. STAR Protoc. 2022 Jan 20;3(1):101100. doi: 10.1016/j.xpro.2021.101100. PMID: 35098158; PMCID: PMC8783141.
[5] Akdogan-Ozdilek B, Duval KL, Meng FW, Murphy PJ, Goll MG. Identification of chromatin states during zebrafish gastrulation using CUT&RUN and CUT&Tag. Dev Dyn. 2022 Apr;251(4):729-742. doi: 10.1002/dvdy.430. Epub 2021 Oct 23. PMID: 34647658; PMCID: PMC8976701.
[6] Tao X, Feng S, Zhao T, Guan X. Efficient chromatin profiling of H3K4me3 modification in cotton using CUT&Tag. Plant Methods. 2020 Aug 31;16:120. doi: 10.1186/s13007-020-00664-8. PMID: 32884577; PMCID: PMC7460760.
.png)

