NGS新品来啦

5R-16S rRNA测序服务
解析肿瘤及其他微环境微生物组的高分辨率新利器
肿瘤等复杂微环境中低丰度微生物(如乳腺癌中的核梭杆菌【1】)具有重要临床意义,但其生物量仅占样本总 DNA 的 0.01%-1%,易受宿主 DNA 干扰。传统检测技术存在明显局限:短读长 16S rRNA 测序(如 V3-V4 区)仅覆盖全长基因的 20%-30%,物种水平鉴定精度不足,且对降解样本(如 FFPE)灵敏度低;宏基因组测序虽覆盖全面,但成本高,在低微生物量样本中效率低下(数据多来源于宿主 DNA)。
针对上述痛点,生工生物创新推出 “5R-16S 多重扩增测序”技术。该技术通过多重 PCR 扩增原核生物 16S rRNA 基因的 5 个关键高变区(V2、V3、V5、V6、V8),显著提升微生物基因覆盖度与分辨率。依托 16S rRNA 基因 “高度保守性 + 关键区域序列变异” 的进化特性,该技术尤其适用于低微生物量样本(如肿瘤组织),可有效克服宿主 DNA 干扰,精准捕获微环境微生物信号。
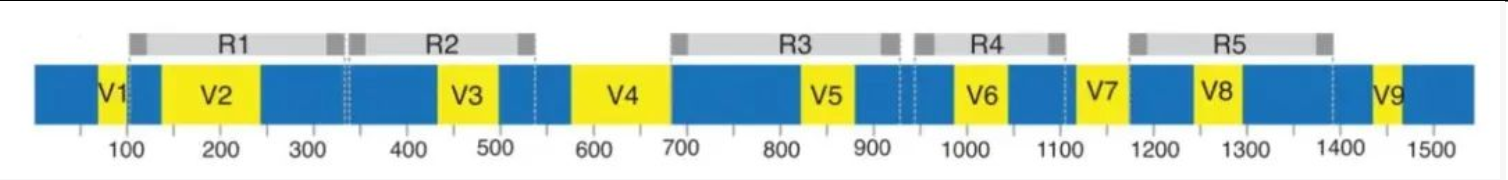 5R-16S 测序检测原理图【2】
5R-16S 测序检测原理图【2】
分析流程
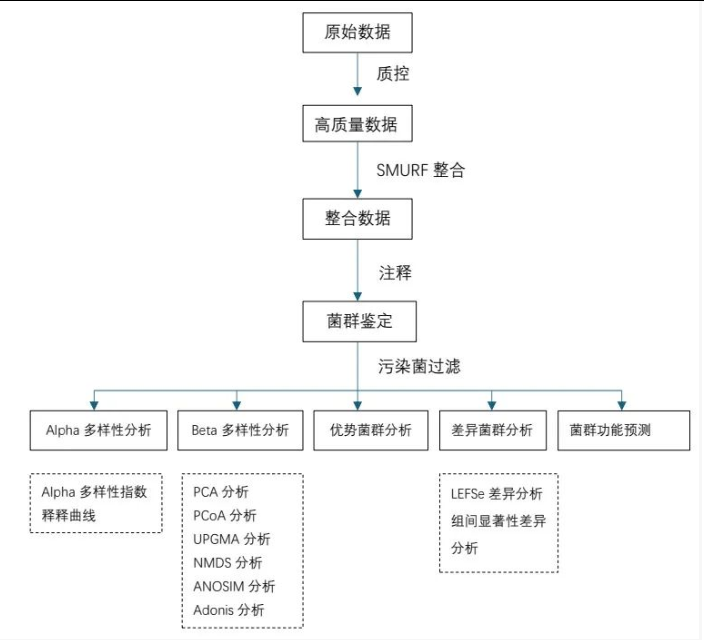
技术分析涵盖多样性散点图、物种差异统计箱线图、LEFSE LDA 图等核心内容,支持微生物群落多样性分析、差异物种鉴定等深度解读。
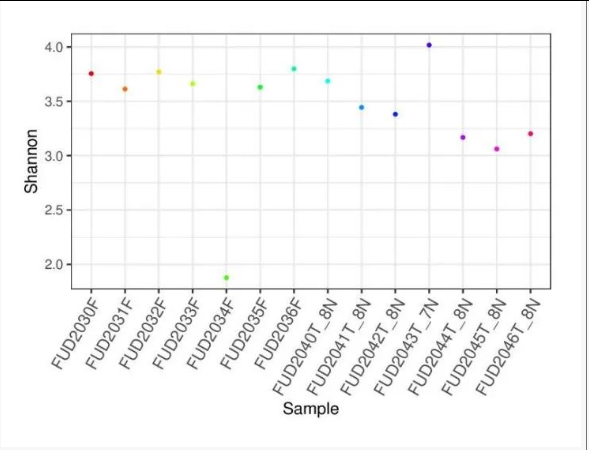
多样性散点图
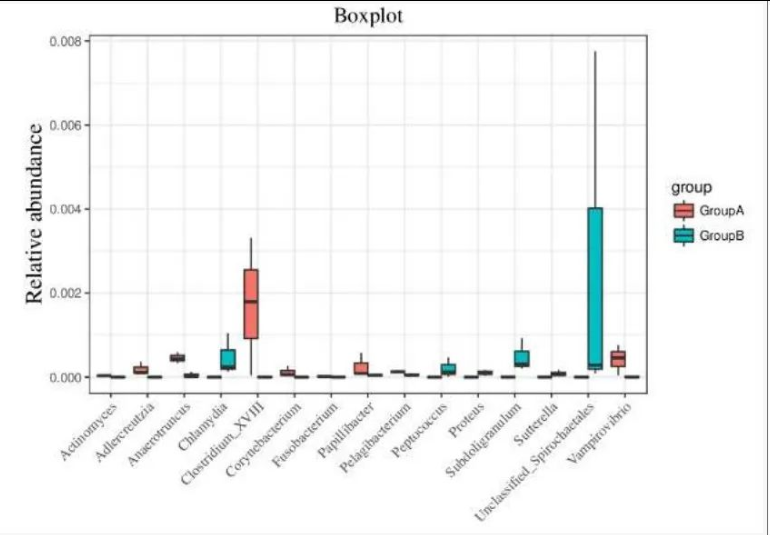
物种差异统计箱线图
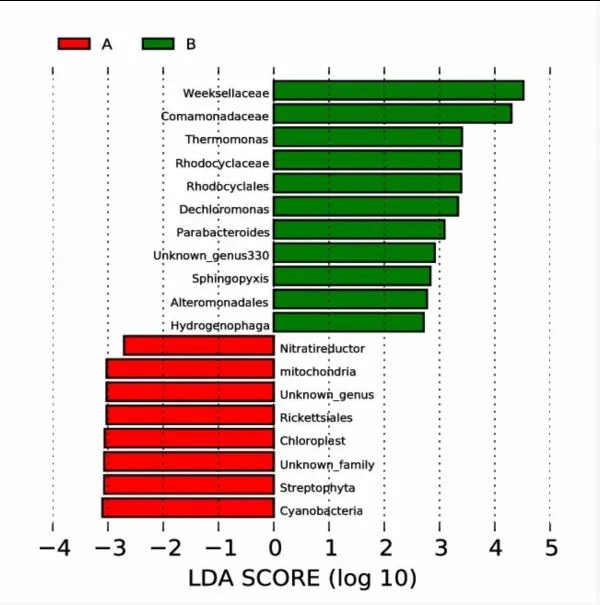
LEFSE LDA图
标题:结直肠癌不同隔室肿瘤细胞相关微生物群的检测与比较
期刊:Frontiers in Oncology(2024, IF≈10.34)【3】

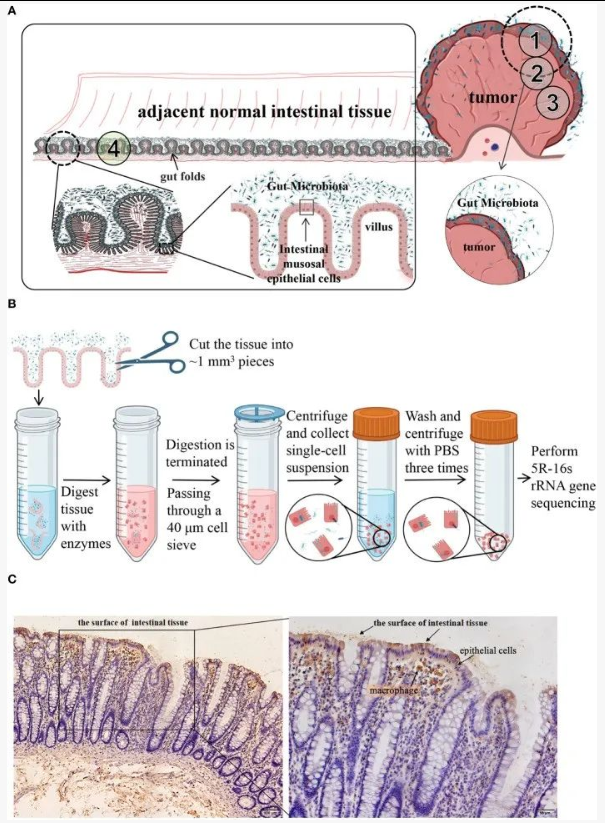
本研究采集11例未使用抗生素的结直肠癌(CRC)患者手术样本,分为四组:
1. TT:肿瘤组织 (Tumor Tissue)
2. NT:癌旁正常组织 (Adjacent Normal Tissue)
3. TC:酶消化分离的肿瘤细胞 (Tumor Cells)
4. NC:酶消化分离的正常细胞 (Normal Cells)
另取 1 例样本验证采样部位(含黏膜层与否)及清洗次数对微生物检测的影响。采用 5R-16S rRNA 测序(覆盖 V2/V3/V5/V6/V8 区),结合 Greengenes 数据库注释,分析包括 Alpha/Beta 多样性、LEfSe 差异物种鉴定、PICRUSt2 功能预测等。
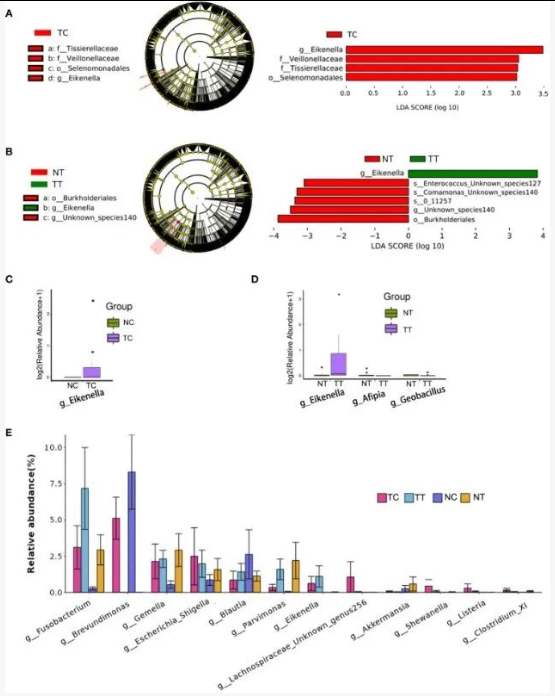
样本处理的影响:采样部位(含黏膜层与否)和清洗次数显著改变微生物组成。未去除黏膜层样本(受粪便污染)以厚壁菌门、拟杆菌门为主;去除黏膜层并充分清洗后,变形菌门、蓝藻门比例升高,普雷沃菌属、粪杆菌属等丰度降低。
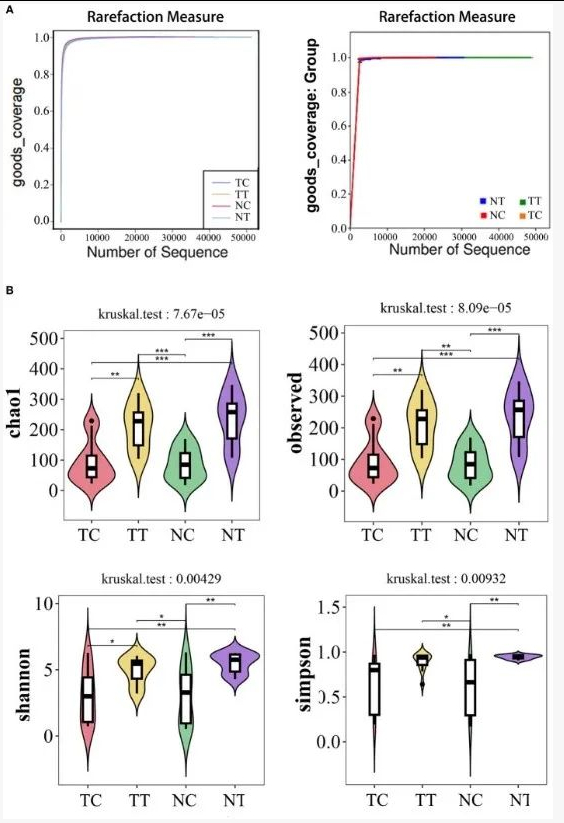
去污染效果:酶消化分离细胞(TC/NC)结合 40 μm 滤网过滤及离心清洗,可显著减少粪便细菌污染;细胞样本(TC/NC)的物种丰富度(Chao1 指数)显著低于组织样本(p<0.05),证实去污染有效。
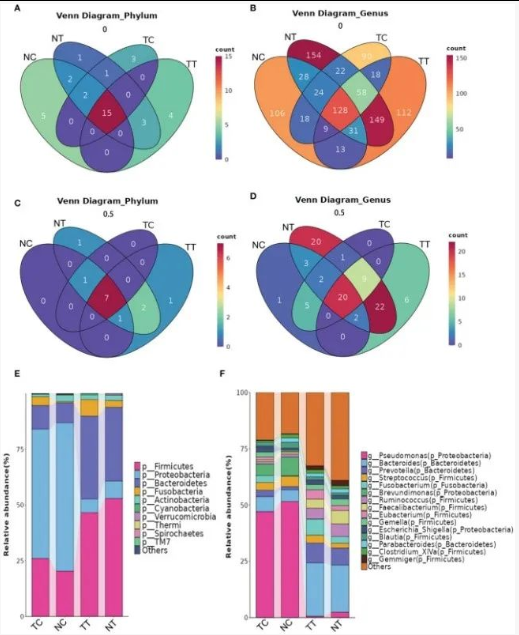
微生物组成差异:细胞样本(TC/NC)与组织样本(TT/NT)微生物组成差异显著 —— 组织样本富集厚壁菌门、拟杆菌门,细胞样本中变形菌门为优势菌门。
肿瘤特异性微生物标志物:
-
肿瘤组织(TT vs NT):核梭杆菌属、艾肯菌属、希瓦氏菌属、李斯特菌属丰度升高;
-
肿瘤细胞(TC vs NC):韦荣球菌科、蒂西菌科丰度升高;
-
正常样本富集:阿克曼菌属在肿瘤样本中丰度降低;
-
细胞 / 组织比例变化:孪生球菌属、埃希氏菌 / 志贺氏菌属、布劳特氏菌属等在细胞样本中比例显著改变。
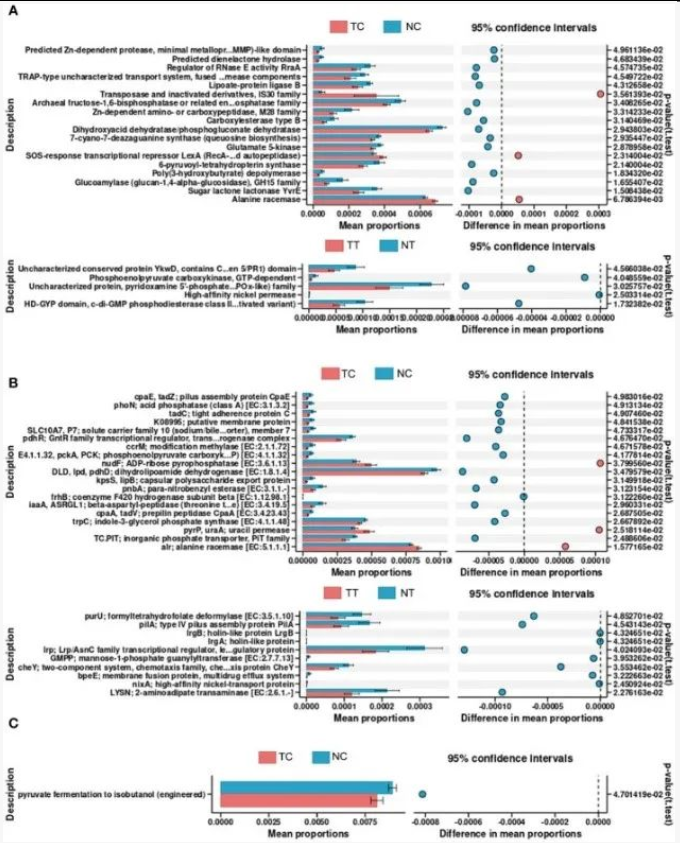
功能差异预测:TC 与 NC 组差异功能基因涉及菌毛组装(如 CpaE)、锌依赖蛋白酶等;TT 与 NT 组差异基因涉及镍转运、c-di-GMP 代谢等。
 |
 |
 |
 |
创新方法有效降低污染:利用酶消化法分离结直肠癌(CRC)肿瘤细胞,结合5R-16S测序,成功获取肿瘤细胞相关微生物群,显著降低了粪便细菌污染。
揭示真实肿瘤内菌群特征:去除污染后,肿瘤细胞样本中的主导菌群由传统组织样本中的厚壁菌门/拟杆菌门转变为变形菌门(Proteobacteria)。
鉴定肿瘤特异性微生物标志物:如核梭杆菌属(Fusobacterium)、艾肯菌属(Eikenella)在肿瘤中显著富集,而阿克曼菌属(Akkermansia)减少。
预测细菌空间分布:整合组织与细胞样本分析,可推断细菌在肿瘤微环境中的定位(例如:某菌在肿瘤组织和肿瘤细胞中均呈现高丰度,提示其可能已侵染至细胞内)。
【1】 Yu T, Guo F, Yu Y, Sun T, Ma D, Han J, Qian Y, Kryczek I, Sun D, Nagarsheth N, Chen Y, Chen H, Hong J, Zou W, Fang JY. Fusobacterium nucleatum Promotes Chemoresistance to Colorectal Cancer by Modulating Autophagy. Cell.
【2】 Nejman D, Livyatan I, Fuks G, Gavert N, Zwang Y, Geller LT, Rotter-Maskowitz A, Weiser R, Mallel G, Gigi E, Meltser A, Douglas GM, Kamer I, Gopalakrishnan V, Dadosh T, Levin-Zaidman S, Avnet S, Atlan T, Cooper ZA, Arora R, Cogdill AP, Khan MAW, Ologun G, Bussi Y, Weinberger A, Lotan-Pompan M, Golani O, Perry G, Rokah M, Bahar-Shany K, Rozeman EA, Blank CU, Ronai A, Shaoul R, Amit A, Dorfman T, Kremer R, Cohen ZR, Harnof S, Siegal T, Yehuda-Shnaidman E, Gal-Yam EN, Shapira H, Baldini N, Langille MGI, Ben-Nun A, Kaufman B, Nissan A, Golan T, Dadiani M, Levanon K, Bar J, Yust-Katz S, Barshack I, Peeper DS, Raz DJ, Segal E, Wargo JA, Sandbank J, Shental N, Straussman R. The human tumor microbiome is composed of tumor type-specific intracellular bacteria.
【3】 Zuo, Y., Lu, Y., Pang, J., Jin, S., Zhang, X., Zhao, E., & Li, Y. (2024). Detection and comparison of tumor cell-associated microbiota from different compartments of colorectal cancer.
如您对5R-16S多重扩增测序项目感兴趣或希望了解更多技术细节,欢迎联系您所在地的生工生物销售办事处,或直接咨询我们的专业技术支持团队。
技术支持
电话:
021-5707 2083 / 2062 / 2064
.png)

