文末一键获取WB实验超全技巧
蛋白糖基化是一种非常重要而且广泛的蛋白质翻译后修饰方式,在所有的已知蛋白质中,约有50%是被糖基化的蛋白质。哺乳动物中的糖链大约有7000+种结构,大大扩展了蛋白质组。糖基化在细胞信号传导、细胞黏附和识别、蛋白稳定性、蛋白功能调节扮演着关键角色。WB也是研究糖基化修饰的常用实验,那么我们在检测的时候应该注意哪些细节呢,今天小优细节君来和大家一起探讨一下。
分子量偏移
糖基化修饰会增大分子量,这个大家都很容易理解,但是不同样本,相同样本不同刺激处理的糖基化增加的分子量并非固定值。同一蛋白上的糖基化位点可能连接不同长度、分支结构和糖基组成的糖链(如高甘露糖型、杂合型、复杂型),导致分子量存在差异。还有大体积糖链可能阻碍SDS与蛋白结合,导致SDS包裹不均匀,迁移速度不一致,呈现出表观分子量不一致。例如下图的MUC1蛋白,属于高度糖基化蛋白,理论分子量只有120kd,但在糖基化修饰后,分子量可达到400kd左右,因为含有大量的O-糖基化位点,导致糖链占比非常大。
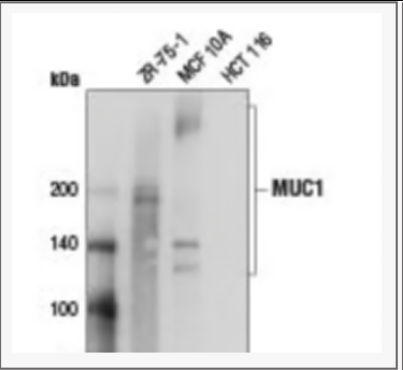
来自CSThttps://www.univ-bio.com/search.html?keyword=14161&mk_channel=WXktzj#14161
下图的EGFR理论分子量是134kd,由于有10+个N-糖基化位点,实际分子量可达到175-180kd,增加~40kd。
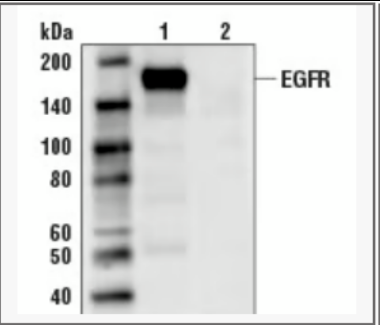
来自CSThttps://www.univ-bio.com/search.html?keyword=4267&mk_channel=WXktzj#4267
所以,我们在检测糖基化修饰的蛋白的时候,一定要把膜裁宽一些,第一次做可以做个全膜,确认条带位置,再进行重复实验。
糖苷酶的选择
单纯依靠分子量偏移不足以确认糖基化,必须设计糖苷酶对照实验。糖苷酶的选择要看糖基的连接方式,例如与Asn侧链的酰胺键相连(N-糖基化),与Ser、Thr、羟基赖氨酸(胶原)或Tyr(糖原蛋白)的羟基相连(O-糖基化),或通过C-C键连接到Trp的C2位置(C-甘露糖基化)。另外,还可以通过糖基磷脂酰肌醇(GPI)锚间接与蛋白质相连。
N-糖基化是真核生物中最普遍的糖基化形式,推荐使用PNGase F,PNGase F处理后的样本应显示分子量降低。如果是O-糖基化,应使用 O-糖苷酶,不过这个酶特异性没有PNGase F高,一般是混合酶,使用前需要对样本进行处理。
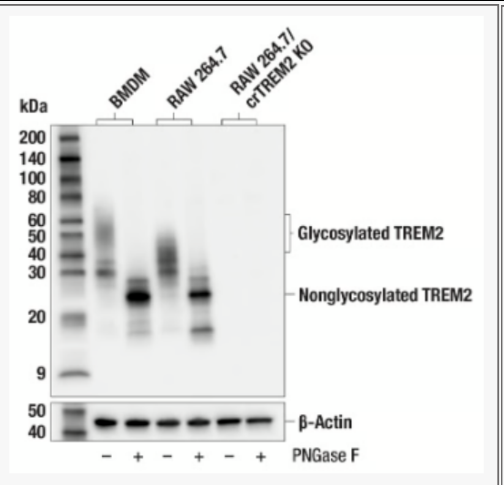
来自CSThttps://www.univ-bio.com/search.html?keyword=52749&mk_channel=WXktzj#52749
有小伙伴要问了,那我怎么知道我的蛋白发生什么糖基化了。这个很简单,我们通过Uniprot-PTM,就可以查看具体发生糖基化的类型了。

条带形状
很多小伙伴问,为什么我的条带是粗粗的,非常模糊,还有拖尾,看起来像样本降解,问怎么优化实验。其实这都是糖基化修饰条带正常的现象,如果条带趋势符合预期,无需再优化。如果条带过粗,可以尝试减少上样量和抗体浓度。如果严重拖尾,我们可以尝试使用梯度胶+低压电泳的办法,尽可能减少拖尾现象。
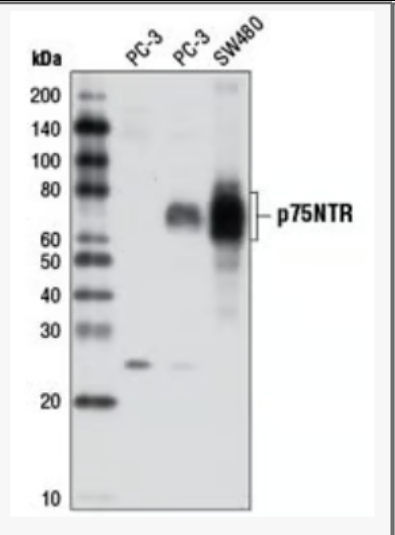
来自CSThttps://www.univ-bio.com/search.html?keyword=8238&mk_channel=WXktzj#8238
样本制备
关于样本制备,网上说法不一,有人说应该充分变性,有人说要不煮样,那么到底应该怎么处理呢,今天我们来讨论一下。
对于高度糖基化的蛋白,糖链非常多,我们确实应该要充分变性,暴露出抗原位点,这样抗体才能结合。可能普通的RIPA裂解液还不够,还需要加SDS或者尿素等充分变性。糖基化修饰的蛋白大部分为膜蛋白,膜蛋白如果高温煮样又容易聚集影响电泳,所以可以尝试低温煮样,或者不煮样,加上超声处理。
有小伙伴要问了,不煮样如何充分变性呢?其实这两点并不矛盾,化学变性也是变性,我们可以适当降低煮样温度(37度或者70度),提高裂解液中的变性成分(尿素等)使蛋白充分变性。还有最重要一点,不同的蛋白需要做预实验来摸索最佳条件,切勿用一个条件来做所有蛋白。
最后,总结一下糖基化WB需要注意的点:
❖
全膜保证目的蛋白不丢失
❖
糖苷酶验证特异性
❖
电泳改善模糊拖尾
❖
样本制备需充分变性
参考文献:
1. Eichler J. Protein glycosylation. Curr Biol. 2019 Apr 1;29(7):R229-R231.
2. Stowell SR, Ju T, Cummings RD. Protein glycosylation in cancer. Annu Rev Pathol. 2015;10:473-510.
📚https://www.univ-bio.com/study-center/literatureList.html?mk_channel=WXktzj点此直达【文献中心】:超百万高质量文献数据,每月5万+持续更新!

